[English] 日本語
 Yorodumi
Yorodumi- PDB-1tnr: CRYSTAL STRUCTURE OF THE SOLUBLE HUMAN 55 KD TNF RECEPTOR-HUMAN T... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1tnr | ||||||
|---|---|---|---|---|---|---|---|
| Title | CRYSTAL STRUCTURE OF THE SOLUBLE HUMAN 55 KD TNF RECEPTOR-HUMAN TNF-BETA COMPLEX: IMPLICATIONS FOR TNF RECEPTOR ACTIVATION | ||||||
 Components Components |
| ||||||
 Keywords Keywords | COMPLEX(LYMPHOKINE/RECEPTOR) / COMPLEX(LYMPHOKINE-RECEPTOR) / COMPLEX(LYMPHOKINE-RECEPTOR) complex | ||||||
| Function / homology |  Function and homology information Function and homology information: / tumor necrosis factor receptor superfamily complex / pulmonary valve development / aortic valve development / positive regulation of chronic inflammatory response to antigenic stimulus / negative regulation of extracellular matrix constituent secretion / tumor necrosis factor receptor activity / positive regulation of apoptotic process involved in morphogenesis / : / tumor necrosis factor binding ...: / tumor necrosis factor receptor superfamily complex / pulmonary valve development / aortic valve development / positive regulation of chronic inflammatory response to antigenic stimulus / negative regulation of extracellular matrix constituent secretion / tumor necrosis factor receptor activity / positive regulation of apoptotic process involved in morphogenesis / : / tumor necrosis factor binding / positive regulation of humoral immune response mediated by circulating immunoglobulin / TNFs bind their physiological receptors / TNF signaling / TNF receptor superfamily (TNFSF) members mediating non-canonical NF-kB pathway / negative regulation of cardiac muscle hypertrophy / regulation of establishment of endothelial barrier / regulation of tumor necrosis factor-mediated signaling pathway / tumor necrosis factor receptor binding / positive regulation of extrinsic apoptotic signaling pathway / TNFR1-induced proapoptotic signaling / TNFR1-mediated ceramide production / prostaglandin metabolic process / positive regulation of lipid metabolic process / Interleukin-10 signaling / humoral immune response / positive regulation of glial cell proliferation / extrinsic apoptotic signaling pathway via death domain receptors / positive regulation of execution phase of apoptosis / cell surface receptor signaling pathway via JAK-STAT / lymph node development / negative regulation of fibroblast proliferation / response to nutrient / cytokine activity / tumor necrosis factor-mediated signaling pathway / protein localization to plasma membrane / TNFR1-induced NF-kappa-B signaling pathway / TNFR2 non-canonical NF-kB pathway / Regulation of TNFR1 signaling / cellular response to mechanical stimulus / negative regulation of inflammatory response / positive regulation of type II interferon production / cytokine-mediated signaling pathway / intrinsic apoptotic signaling pathway in response to DNA damage / positive regulation of inflammatory response / cell-cell signaling / signaling receptor activity / response to lipopolysaccharide / transcription by RNA polymerase II / response to hypoxia / cell surface receptor signaling pathway / positive regulation of canonical NF-kappaB signal transduction / receptor complex / defense response to bacterium / defense response to Gram-positive bacterium / immune response / membrane raft / response to xenobiotic stimulus / inflammatory response / Golgi membrane / signaling receptor binding / apoptotic process / cell surface / signal transduction / positive regulation of transcription by RNA polymerase II / extracellular space / extracellular region / membrane / plasma membrane Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / Resolution: 2.85 Å X-RAY DIFFRACTION / Resolution: 2.85 Å | ||||||
 Authors Authors | Banner, D.W. | ||||||
 Citation Citation |  Journal: Cell(Cambridge,Mass.) / Year: 1993 Journal: Cell(Cambridge,Mass.) / Year: 1993Title: Crystal structure of the soluble human 55 kd TNF receptor-human TNF beta complex: implications for TNF receptor activation. Authors: Banner, D.W. / D'Arcy, A. / Janes, W. / Gentz, R. / Schoenfeld, H.J. / Broger, C. / Loetscher, H. / Lesslauer, W. #1: Journal: J.Mol.Biol. / Year: 1993 Title: Crystallization and preliminary crystallographic analysis of a TNF-beta-55 kDa TNF receptor complex. Authors: D'Arcy, A. / Banner, D.W. / Janes, W. / Winkler, F.K. / Loetscher, H. / Schonfeld, H.J. / Zulauf, M. / Gentz, R. / Lesslauer, W. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1tnr.cif.gz 1tnr.cif.gz | 68.4 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1tnr.ent.gz pdb1tnr.ent.gz | 51.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1tnr.json.gz 1tnr.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/tn/1tnr https://data.pdbj.org/pub/pdb/validation_reports/tn/1tnr ftp://data.pdbj.org/pub/pdb/validation_reports/tn/1tnr ftp://data.pdbj.org/pub/pdb/validation_reports/tn/1tnr | HTTPS FTP |
|---|
-Related structure data
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
|---|
- Links
Links
- Assembly
Assembly
| Deposited unit | 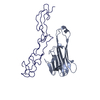
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 15872.904 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Production host: Homo sapiens (human) / Production host:  |
|---|---|
| #2: Protein | Mass: 15766.797 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Production host: SF9 cells / References: UniProt: P19438 Homo sapiens (human) / Production host: SF9 cells / References: UniProt: P19438 |
| #3: Water | ChemComp-HOH / |
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION X-RAY DIFFRACTION |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 4.07 Å3/Da / Density % sol: 69.75 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | *PLUS pH: 5.5 / Method: vapor diffusion, hanging drop | ||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Reflection | *PLUS Highest resolution: 2.83 Å / Num. obs: 13270 / % possible obs: 99.7 % / Num. measured all: 43171 / Rmerge(I) obs: 0.078 |
|---|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 2.85→50 Å / σ(F): 2 Details: THE RECEPTOR REGIONS R 15 - R 29 AND R 144 - R 153, I.E. THE N- AND C-TERMINAL ENDS OF THE RECEPTOR, MUST BE REGARDED AS TENTATIVE. THERE IS WEAK ELECTRON DENSITY FOR THE THREE CARBOHYDRATE ...Details: THE RECEPTOR REGIONS R 15 - R 29 AND R 144 - R 153, I.E. THE N- AND C-TERMINAL ENDS OF THE RECEPTOR, MUST BE REGARDED AS TENTATIVE. THERE IS WEAK ELECTRON DENSITY FOR THE THREE CARBOHYDRATE CHAINS ATTACHED TO ASPARAGINES R 25, R 116 AND R 122. THESE POSITIONS HAVE BEEN MARKED WITH WATER MOLECULES 45, 42 AND 43, RESPECTIVELY. ASPARTIC ACID A 50 ADOPTS THE UNUSUAL EPSILON CONFORMATION ON BINDING THE RECEPTOR. THE PEPTIDE PLANES A 40 - A 41 AND R 100 - R 101 MAY HAVE THE WRONG ORIENTATION.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.85→50 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Software | *PLUS Name:  X-PLOR / Classification: refinement X-PLOR / Classification: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement | *PLUS Rfactor obs: 0.16 / Rfactor Rwork: 0.16 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints | *PLUS Type: x_angle_d / Dev ideal: 1.8 |
 Movie
Movie Controller
Controller


 PDBj
PDBj









