+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6asb | ||||||
|---|---|---|---|---|---|---|---|
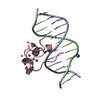
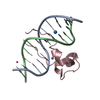
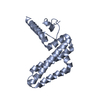
| タイトル | CXXC and PHD-type zinc finger regions of FBXL19 in complex with DNA | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | DNA BINDING PROTEIN/DNA / CXXC zinc finger / PHD zinc finger / Structural Genomics / Structural Genomics Consortium / SGC / DNA BINDING PROTEIN-DNA complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報unmethylated CpG binding / SCF ubiquitin ligase complex / histone demethylase activity / ubiquitin-like ligase-substrate adaptor activity / transcription coregulator activity / Antigen processing: Ubiquitination & Proteasome degradation / Neddylation / proteasome-mediated ubiquitin-dependent protein catabolic process / regulation of transcription by RNA polymerase II / zinc ion binding ...unmethylated CpG binding / SCF ubiquitin ligase complex / histone demethylase activity / ubiquitin-like ligase-substrate adaptor activity / transcription coregulator activity / Antigen processing: Ubiquitination & Proteasome degradation / Neddylation / proteasome-mediated ubiquitin-dependent protein catabolic process / regulation of transcription by RNA polymerase II / zinc ion binding / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)synthetic construct (人工物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.85 Å 分子置換 / 解像度: 2.85 Å | ||||||
 データ登録者 データ登録者 | Liu, K. / Tempel, W. / Walker, J.R. / Arrowsmith, C.H. / Bountra, C. / Edwards, A.M. / Min, J. / Structural Genomics Consortium (SGC) | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2018 ジャーナル: Structure / 年: 2018タイトル: DNA Sequence Recognition of Human CXXC Domains and Their Structural Determinants. 著者: Xu, C. / Liu, K. / Lei, M. / Yang, A. / Li, Y. / Hughes, T.R. / Min, J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6asb.cif.gz 6asb.cif.gz | 277 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6asb.ent.gz pdb6asb.ent.gz | 217.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6asb.json.gz 6asb.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6asb_validation.pdf.gz 6asb_validation.pdf.gz | 482 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6asb_full_validation.pdf.gz 6asb_full_validation.pdf.gz | 485 KB | 表示 | |
| XML形式データ |  6asb_validation.xml.gz 6asb_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  6asb_validation.cif.gz 6asb_validation.cif.gz | 25.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/as/6asb https://data.pdbj.org/pub/pdb/validation_reports/as/6asb ftp://data.pdbj.org/pub/pdb/validation_reports/as/6asb ftp://data.pdbj.org/pub/pdb/validation_reports/as/6asb | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
| #1: DNA鎖 | 分子量: 3663.392 Da / 分子数: 8 / 由来タイプ: 合成 / 詳細: synthetic DNA / 由来: (合成) synthetic construct (人工物) #2: タンパク質 | 分子量: 13747.133 Da / 分子数: 4 Fragment: CXXC and PHD-type zinc finger regions (UNP residues 31-153) 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: FBXL19, FBL19 / プラスミド: pET28-MHL / 発現宿主: Homo sapiens (ヒト) / 遺伝子: FBXL19, FBL19 / プラスミド: pET28-MHL / 発現宿主:  #3: 化合物 | ChemComp-ZN / |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.61 Å3/Da / 溶媒含有率: 52.96 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法 / pH: 6.5 / 詳細: 28% PEG2000-MME, 0.1 M Bis-Tris |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 1.2831 Å / ビームライン: 19-ID / 波長: 1.2831 Å | ||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2013年2月11日 | ||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1.2831 Å / 相対比: 1 | ||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.85→47.91 Å / Num. obs: 19440 / % possible obs: 97.3 % / 冗長度: 3.7 % / Biso Wilson estimate: 84.61 Å2 / CC1/2: 0.993 / Rmerge(I) obs: 0.1 / Rpim(I) all: 0.061 / Rrim(I) all: 0.117 / Net I/σ(I): 11.8 / Num. measured all: 71758 / Scaling rejects: 0 | ||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: coordinates related to pdb entries 4HP3, 4O64 解像度: 2.85→47.91 Å / Cor.coef. Fo:Fc: 0.895 / Cor.coef. Fo:Fc free: 0.862 / Rfactor Rfree error: 0 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 3.453 / SU Rfree Blow DPI: 0.352 詳細: chainsaw was used to modify the amino acid sequences of models during molecular replacement and refinement. arp/warp was used for the improvement of electron density maps. FBXL19 backbone ...詳細: chainsaw was used to modify the amino acid sequences of models during molecular replacement and refinement. arp/warp was used for the improvement of electron density maps. FBXL19 backbone geometry was restrained to coordinates from PDB entry 4O64. phenix and refmac were used during intermediate refinement stages. restraints for the refinement of metal ion sites were derived from MOGUL queries of CSD structures. coot was used for interactive model building. Model geometry was assessed on the molprobity server.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 212.3 Å2 / Biso mean: 70.64 Å2 / Biso min: 22.87 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.85→47.91 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.85→3 Å / Total num. of bins used: 10
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj













































