+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7b1s | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
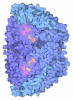
| タイトル | Crystal structure of the ethyl-coenzyme M reductase from Candidatus Ethanoperedens thermophilum at 0.994-A resolution | |||||||||
 要素 要素 | (Ethyl-Coenzyme M reductase ...) x 3 | |||||||||
 キーワード キーワード | TRANSFERASE / Ethyl-CoM reductase / Methyl-CoM reductase / ethane-oxidizers / F430-cofactor / post-translational modification / gas channel / coenzyme M / coenzyme B / true atomic resolution / thermophile / archaea. | |||||||||
| 機能・相同性 | 1-THIOETHANESULFONIC ACID / : / : / Coenzyme B / Dimethylated-F430 cofactor / Chem-UUT 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 0.992 Å 分子置換 / 解像度: 0.992 Å | |||||||||
 データ登録者 データ登録者 | Wagner, T. / Lemaire, O.N. / Engilberge, S. | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Crystal structure of a key enzyme for anaerobic ethane activation. 著者: Hahn, C.J. / Lemaire, O.N. / Kahnt, J. / Engilberge, S. / Wegener, G. / Wagner, T. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7b1s.cif.gz 7b1s.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7b1s.ent.gz pdb7b1s.ent.gz | 975.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7b1s.json.gz 7b1s.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b1/7b1s https://data.pdbj.org/pub/pdb/validation_reports/b1/7b1s ftp://data.pdbj.org/pub/pdb/validation_reports/b1/7b1s ftp://data.pdbj.org/pub/pdb/validation_reports/b1/7b1s | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Ethyl-Coenzyme M reductase ... , 3種, 6分子 ADBECF
| #1: タンパク質 | 分子量: 66344.344 Da / 分子数: 2 / 由来タイプ: 天然 詳細: Seven post-translational modifications exist in this subunit: N1-methylhistidine291 5(S)-methylarginine305 S-methylcysteine354 3-methylisoleucine377 2(S)-methylglutamine445 Thioglycine490 N2-methylhistidine491 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Enrichment culture / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 49874.453 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Enrichment culture / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase #3: タンパク質 | 分子量: 30578.789 Da / 分子数: 2 / 由来タイプ: 天然 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Enrichment culture / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase |
|---|
-非ポリマー , 10種, 3111分子 






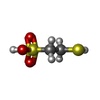











| #4: 化合物 | ChemComp-GOL / #5: 化合物 | #6: 化合物 | ChemComp-MN / | #7: 化合物 | ChemComp-K / #8: 化合物 | ChemComp-CL / #9: 化合物 | #10: 化合物 | #11: 化合物 | #12: 化合物 | #13: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.27 Å3/Da / 溶媒含有率: 45.87 % / 解説: Yellow brick of 250 um long. |
|---|---|
| 結晶化 | 温度: 293.15 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: Crystals were obtained by initial screening at 20 degree celsius using the sitting drop method on a 96-Well MRC 2-Drop Crystallization Plates in polystyrene (SWISSCI). The crystallization ...詳細: Crystals were obtained by initial screening at 20 degree celsius using the sitting drop method on a 96-Well MRC 2-Drop Crystallization Plates in polystyrene (SWISSCI). The crystallization reservoir contained 90 ul of mother liquor, crystallization drop contained a mixture of 0.6 ul protein at 16.22 mg.ml-1 and 0.6 ul precipitant. The crystal was obtained by initial screening using the JBScreen Pentaerythritol screen from Jena Bioscience in a Coy tent under an N2/H2 atmosphere (95:5 %). The crystallization reservoir contained 45 % (w/v) Pentaerythritol Propoxylate (5/4 PO/OH), 100 mM Tris pH 8.5 and 400 mM potassium chloride. PH範囲: 7.5 - 8.5 / Temp details: / |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06DA / 波長: 1.00003 Å / ビームライン: X06DA / 波長: 1.00003 Å |
| 検出器 | タイプ: DECTRIS PILATUS 2M-F / 検出器: PIXEL / 日付: 2019年10月8日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.00003 Å / 相対比: 1 |
| 反射 | 解像度: 0.992→108.19 Å / Num. obs: 1061996 / % possible obs: 89.1 % / 冗長度: 7 % / CC1/2: 0.997 / Rmerge(I) obs: 0.116 / Rpim(I) all: 0.047 / Rrim(I) all: 0.125 / Net I/σ(I): 9.7 |
| 反射 シェル | 解像度: 0.992→1.07 Å / Rmerge(I) obs: 1.167 / Mean I/σ(I) obs: 1.6 / Num. unique obs: 53103 / CC1/2: 0.563 / Rpim(I) all: 0.476 / Rrim(I) all: 1.262 / % possible all: 58.1 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5A8W 解像度: 0.992→39.76 Å / SU ML: 0.06 / 交差検証法: THROUGHOUT / σ(F): 1.35 / 位相誤差: 11.99 / 立体化学のターゲット値: ML 詳細: Refinement was performed with hydrogens in riding positions
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 49.64 Å2 / Biso mean: 10.6019 Å2 / Biso min: 3.16 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 0.992→39.76 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 30
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj