[English] 日本語
 Yorodumi
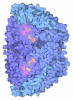
Yorodumi- PDB-7nkg: Methyl-coenzyme M reductase from Methermicoccus shengliensis at 1... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7nkg | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Methyl-coenzyme M reductase from Methermicoccus shengliensis at 1.6-A resolution | ||||||||||||
 Components Components | (Methyl-coenzyme M reductase ...) x 3 | ||||||||||||
 Keywords Keywords | TRANSFERASE / Methyl-coenzyme M reductase / cofactor F430 / post-translational modification / archaea / methoxydotrophy / methanogenesis / coenzyme M / coenzyme B / heterodisulfide / radical mechanism / thermophile / thioglycine | ||||||||||||
| Function / homology | 1-THIOETHANESULFONIC ACID / FACTOR 430 / : / Coenzyme B Function and homology information Function and homology information | ||||||||||||
| Biological species |  Methermicoccus shengliensis DSM 18856 (archaea) Methermicoccus shengliensis DSM 18856 (archaea) | ||||||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.6 Å MOLECULAR REPLACEMENT / Resolution: 1.6 Å | ||||||||||||
 Authors Authors | Mueller, M. / Wagner, T. | ||||||||||||
| Funding support |  Germany, Germany,  Netherlands, 3items Netherlands, 3items
| ||||||||||||
 Citation Citation |  Journal: Microorganisms / Year: 2021 Journal: Microorganisms / Year: 2021Title: Structural Insights into the Methane-Generating Enzyme from a Methoxydotrophic Methanogen Reveal a Restrained Gallery of Post-Translational Modifications. Authors: Kurth, J.M. / Muller, M.C. / Welte, C.U. / Wagner, T. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7nkg.cif.gz 7nkg.cif.gz | 1.9 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7nkg.ent.gz pdb7nkg.ent.gz | 1.6 MB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7nkg.json.gz 7nkg.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/nk/7nkg https://data.pdbj.org/pub/pdb/validation_reports/nk/7nkg ftp://data.pdbj.org/pub/pdb/validation_reports/nk/7nkg ftp://data.pdbj.org/pub/pdb/validation_reports/nk/7nkg | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1e6yS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Methyl-coenzyme M reductase ... , 3 types, 12 molecules ADGJBEHKCFIL
| #1: Protein | Mass: 62295.250 Da / Num. of mol.: 4 / Mutation: wild-type / Source method: isolated from a natural source Details: The polypeptide contains three post-translational modifications: Methylhistidine-275 Methylarginine-289 Thioglycine-463 Source: (natural)  Methermicoccus shengliensis DSM 18856 (archaea) Methermicoccus shengliensis DSM 18856 (archaea)Cell line: / / Organ: / / Plasmid details: / / Variant: / / Strain: ZC-1 / Tissue: / / References: coenzyme-B sulfoethylthiotransferase #2: Protein | Mass: 45908.246 Da / Num. of mol.: 4 / Mutation: wild-type / Source method: isolated from a natural source Source: (natural)  Methermicoccus shengliensis DSM 18856 (archaea) Methermicoccus shengliensis DSM 18856 (archaea)Cell line: / / Organ: / / Plasmid details: / / Variant: / / Strain: ZC-1 / Tissue: / / References: coenzyme-B sulfoethylthiotransferase #3: Protein | Mass: 28124.594 Da / Num. of mol.: 4 / Mutation: wild-type / Source method: isolated from a natural source Source: (natural)  Methermicoccus shengliensis DSM 18856 (archaea) Methermicoccus shengliensis DSM 18856 (archaea)Cell line: / / Organ: / / Plasmid details: / / Variant: / / Strain: ZC-1 / Tissue: / / References: coenzyme-B sulfoethylthiotransferase |
|---|
-Non-polymers , 7 types, 4320 molecules 

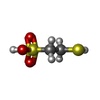

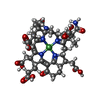








| #4: Chemical | | #5: Chemical | #6: Chemical | ChemComp-COM / #7: Chemical | ChemComp-TP7 / #8: Chemical | ChemComp-F43 / #9: Chemical | ChemComp-SO4 / #10: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.12 Å3/Da / Density % sol: 42.12 % Description: Yellow thick brick-shape crystals appeared within two weeks |
|---|---|
| Crystal grow | Temperature: 291.15 K / Method: vapor diffusion, sitting drop / pH: 5.5 Details: The protein sample was at 47 g/l in 25 mM Tris/HCl pH 7.6, 10% v/v glycerol and 2 mM dithiothreitol. MCR crystals were obtained aerobically by using the sitting drop method on 96-Well MRC 2- ...Details: The protein sample was at 47 g/l in 25 mM Tris/HCl pH 7.6, 10% v/v glycerol and 2 mM dithiothreitol. MCR crystals were obtained aerobically by using the sitting drop method on 96-Well MRC 2-Drop Crystallization Plates in polystyrene (SWISSCI). The crystallization reservoir contained 90 ul of mother liquor; the crystallization drop contained a mixture of 0.6 ul protein sample and 0.6 ul of crystallization solution. The crystallization solution contained 25% w/v polyethylene glycol 3350, 100 mM Bis-Tris pH 5.5 and 200 mM lithium sulfate. PH range: / / Temp details: / |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SOLEIL SOLEIL  / Beamline: PROXIMA 1 / Wavelength: 0.97856 Å / Beamline: PROXIMA 1 / Wavelength: 0.97856 Å |
| Detector | Type: DECTRIS EIGER X 16M / Detector: PIXEL / Date: Nov 23, 2019 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97856 Å / Relative weight: 1 |
| Reflection | Resolution: 1.6→49.41 Å / Num. obs: 602614 / % possible obs: 99.7 % / Redundancy: 4.2 % / CC1/2: 0.997 / Rmerge(I) obs: 0.091 / Rpim(I) all: 0.051 / Rrim(I) all: 0.105 / Net I/σ(I): 8.3 |
| Reflection shell | Resolution: 1.6→1.69 Å / Redundancy: 4.3 % / Rmerge(I) obs: 1.216 / Mean I/σ(I) obs: 1 / Num. unique obs: 87124 / CC1/2: 0.356 / Rpim(I) all: 0.661 / Rrim(I) all: 1.388 / % possible all: 99.3 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 1E6Y Resolution: 1.6→48.36 Å / Cor.coef. Fo:Fc: 0.963 / Cor.coef. Fo:Fc free: 0.957 / SU R Cruickshank DPI: 0.107 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.09 / SU Rfree Blow DPI: 0.083 / SU Rfree Cruickshank DPI: 0.081 Details: The last cycle of refinement was performed with hydrogens in riding positions
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 136.35 Å2 / Biso mean: 42.85 Å2 / Biso min: 12.79 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.21 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 1.6→48.36 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.6→1.61 Å / Rfactor Rfree error: 0 / Total num. of bins used: 51
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj