-検索条件
-検索結果
検索 (著者・登録者: zhu & yl)の結果102件中、1から50件目までを表示しています

EMDB-37727: 
Cryo-ET structure of RuBisCO from 3.9 angstroms Synechococcus elongatus PCC 7942

EMDB-37728: 
Cryo-ET map of RuBisCO at 4.4 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome
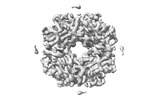
EMDB-37729: 
Cryo-ET map of RuBisCO-SSUL at 5.9 angstroms from Synechococcus elongatus PCC 7942 beta-carboxysome

EMDB-37730: 
Cryo-ET map of RuBisCO at the outermost layer that is loosely attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome

EMDB-37731: 
Cryo-ET map of RuBisCO at the outermost layer that is tightly attached to the shell of Synechococcus elongatus PCC 7942 beta-carboxysome
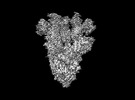
EMDB-28198: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with LLNL-199

EMDB-28199: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with 2130-1-0114-112

PDB-8ekd: 
Cryo-EM map of SARS-CoV-2 Omicron BA.2 spike in complex with 2130-1-0114-112

EMDB-41725: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-41727: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyl: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

PDB-8tyo: 
Structural and biochemical rationale for Beta variant protein booster vaccine broad cross-neutralization of SARS-CoV-2

EMDB-33650: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7

EMDB-33651: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 (focused refinement on Fab-RBD interface)

PDB-7y71: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7

PDB-7y72: 
SARS-CoV-2 spike glycoprotein trimer complexed with Fab fragment of anti-RBD antibody E7 (focused refinement on Fab-RBD interface)

EMDB-29735: 
Structure of nucleosome-bound Sirtuin 6 deacetylase

PDB-8g57: 
Structure of nucleosome-bound Sirtuin 6 deacetylase

EMDB-34678: 
Cyanophage Pam3 neck
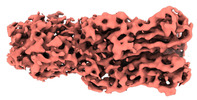
EMDB-33799: 
Cyanophage Pam3 fiber
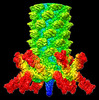
EMDB-33802: 
Cyanophage Pam3 baseplate proteins

EMDB-34679: 
Cyanophage Pam3 portal-adaptor

EMDB-34680: 
Cyanophage Pam3 capsid asymmetric unit

EMDB-34681: 
Cyanophage Pam3 Sheath-tube

EMDB-33386: 
Structure of Apo-hSLC19A1

EMDB-33387: 
Structure of hSLC19A1+3'3'-CDA

EMDB-33388: 
Structure of hSLC19A1+2'3'-CDAS

EMDB-33389: 
Structure of hSLC19A1+2'3'-cGAMP

EMDB-34176: 
Structure of hSLC19A1+5-MTHF

EMDB-34177: 
Structure of hSLC19A1+PMX

EMDB-26636: 
E.coli RNaseP Holoenzyme with Mg2+
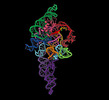
EMDB-26637: 
E.coli RNaseP Holoenzyme with Mg2+

EMDB-26638: 
E.coli RNaseP Holoenzyme with Mg2+
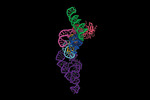
EMDB-26640: 
E.coli RNaseP Holoenzyme with Mg2+

PDB-7uo0: 
E.coli RNaseP Holoenzyme with Mg2+

PDB-7uo1: 
E.coli RNaseP Holoenzyme with Mg2+

PDB-7uo2: 
E.coli RNaseP Holoenzyme with Mg2+

PDB-7uo5: 
E.coli RNaseP Holoenzyme with Mg2+

EMDB-26507: 
SARS-CoV-2 spike in complex with Multivalent miniprotein inhibitor FUS231-P24 (2RBDs open)

EMDB-26508: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS231-P24 (3RBDs open)

EMDB-26509: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (2RBDs open)

EMDB-26510: 
SARS-CoV-2 spike in complex with multivalent miniprotein inhibitor FUS31-G10 (3RBDs open)

EMDB-26511: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175 (local refinement of the RBD and AHB2)

EMDB-26512: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175

PDB-7uhb: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175 (local refinement of the RBD and AHB2)

PDB-7uhc: 
SARS-CoV-2 spike in complex with AHB2-2GS-SB175

EMDB-25785: 
SARS-CoV-2 spike in complex with the S2K146 neutralizing antibody Fab fragment (three receptor-binding domains open)

EMDB-25783: 
SARS-CoV-2 spike in complex with the S2K146 neutralizing antibody Fab fragment (local refinement of the RBD and S2K146)

EMDB-25784: 
SARS-CoV-2 spike in complex with the S2K146 neutralizing antibody Fab fragment (two receptor-binding domains open)

PDB-7tas: 
SARS-CoV-2 spike in complex with the S2K146 neutralizing antibody Fab fragment (local refinement of the RBD and S2K146)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
