-検索条件
-検索結果
検索 (著者・登録者: zhang & yx)の結果72件中、1から50件目までを表示しています

EMDB-60254: 
Vesamicol-bound VAChT

EMDB-60255: 
Acetylcholine-bound VAChT

EMDB-39107: 
SARS-CoV-2 DMV nsp3-4 pore complex (full-pore)

EMDB-39109: 
SARS-CoV-2 DMV nsp3-4 pore complex (consensus-pore, C6 symmetry)

EMDB-39111: 
SARS-CoV-2 DMV nsp3-4 pore complex (extended-pore)

EMDB-39112: 
SARS-CoV-2 DMV nsp3-4 pore complex (consensus-pore, C3 symmetry)

EMDB-39113: 
SARS-CoV-2 DMV nsp3-4 pore complex (mini-pore)

EMDB-39159: 
SARS-CoV-2 DMV nsp3-4 pore complex (full-length-pore)

EMDB-35461: 
Protomer 1 and 2 of the asymmetry trimer of the Cul2-Rbx1-EloBC-FEM1B ubiquitin ligase complex

EMDB-36182: 
An asymmetry dimer of the Cul2-Rbx1-EloBC-FEM1B ubiquitin ligase complexed with BEX2

EMDB-36183: 
Cryo-EM structure of neddylated Cul2-Rbx1-EloBC-FEM1B complexed with FNIP1-FLCN
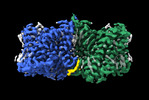
EMDB-36448: 
Structure of AE2 in complex with PIP2
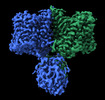
EMDB-36449: 
Structure of R932A/K1147A/H1148A mutant AE2

EMDB-33990: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAbs 3E8 and 5E3 (localized refinement)
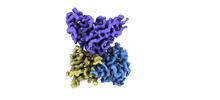
EMDB-33992: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 10E4 (localized refinement)
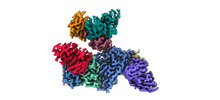
EMDB-33993: 
Cryo-EM density map of EBV gHgL-gp42 in complex with four mAbs 5E3, 3E8, 6H2 and 10E4
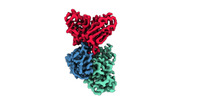
EMDB-33994: 
Cryo-EM structure of EBV gHgL-gp42 in complex with mAb 6H2 (localized refinement)

EMDB-34929: 
Monkeypox virus DNA replication holoenzyme F8, A22 and E4 complex in a DNA binding form

EMDB-34927: 
Cryo-EM structure of monkeypox virus DNA replication holoenzyme F8, A22 and E4 complex without DNA at 2.76 angostram

EMDB-29553: 
Human TMEM175-LAMP1 full-length complex

EMDB-29572: 
Human TMEM175-LAMP1 transmembrane domain only complex

EMDB-32356: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine

EMDB-33310: 
Cryo-EM structure of CopC-CaM-caspase-3 with NAD+

EMDB-33311: 
Cryo-EM structure of CopC-CaM-caspase-3 with ADPR

EMDB-33312: 
Cryo-EM structure of CopC-CaM-caspase-3 with ADPR-deacylization

EMDB-26313: 
C6-guano bound Mu Opioid Receptor-Gi Protein Complex

EMDB-32088: 
Computational design of a potent Epstein-Barr virus fusion protein gB nanoparticle vaccine
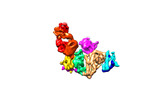
EMDB-33102: 
Cryo-EM structure of EBV glycoprotein complex gHgL-gp42 bound by a neutralizing antibody 6H2

EMDB-33098: 
Structure of somatostatin receptor 2 bound with SST14.

EMDB-33099: 
Structure of somatostatin receptor 2 bound with octreotide.

EMDB-33100: 
Structure of somatostatin receptor 2 bound with lanreotide.

EMDB-32949: 
Structure of Thyrotropin-Releasing Hormone Receptor bound with Taltirelin.

EMDB-32950: 
Structure of Thyrotropin-Releasing Hormone Receptor bound with an Endogenous Peptide Agonist TRH.

EMDB-26314: 
C5guano-uOR-Gi-scFv16

EMDB-32557: 
SARS-CoV-2 Omicron S-open

EMDB-32170: 
SARS-CoV-2 Beta variant spike protein in transition state

EMDB-31146: 
Coupling of N7-methyltransferase and 3'-5' exoribonuclease with SARS-CoV-2 polymerase reveals mechanisms for capping and proofreading

EMDB-31138: 
Co-transcriptional capping machineries in SARS-CoV-2 RTC: Coupling of N7-methyltransferase and 3'-5' exoribonuclease with polymerase reveals mechanisms for capping and proofreading

EMDB-30681: 
Cryo-EM structure of the partial agonist salbutamol-bound beta2 adrenergic receptor-Gs protein complex.

EMDB-30682: 
Cryo-EM structure of the full agonist isoprenaline-bound beta2 adrenergic receptor-Gs protein complex.

EMDB-21249: 
Cryo-EM structure of an activated VIP1 receptor-G protein complex

EMDB-30249: 
Cryo-EM structure of the formoterol-bound beta2 adrenergic receptor-Gs protein complex.

EMDB-20558: 
Atomic Structure of the Human Herpesvirus 6B Capsid and Capsid-Associated Tegument Complexes

EMDB-20559: 
Atomic Structure of the Human Herpesvirus 6B Capsid and Capsid-Associated Tegument Complexes

EMDB-20560: 
Atomic Structure of the Human Herpesvirus 6B Capsid and Capsid-Associated Tegument Complexes

EMDB-20416: 
Cryo-EM structure of the full-length Bacillus subtilis glyQS T-box riboswitch in complex with tRNA-Gly

EMDB-20581: 
In situ structure of BmCPV RNA dependent RNA polymerase at quiescent state

EMDB-20582: 
In situ structure of BmCPV RNA dependent RNA polymerase at initiation state

EMDB-20585: 
In situ structure of BmCPV RNA-dependent RNA polymerase at abortive state

EMDB-20586: 
In situ structure of BmCPV RNA-dependent RNA polymerase at early-elongation state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
