-検索条件
-検索結果
検索 (著者・登録者: spence & j)の結果110件中、1から50件目までを表示しています

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

PDB-8sx3: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10

EMDB-18207: 
Ubiquitin ligation to substrate by a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB-Sil1 peptide, Glacios map

EMDB-18230: 
Ubiquitin ligation to substrate by a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB-Sil1 peptide
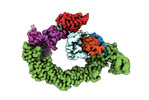
EMDB-18915: 
Ubiquitin ligation to neosubstrate by a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-VHL-MZ1 with trapped UBE2R2~donor UB-BRD4 BD2

PDB-8q7r: 
Ubiquitin ligation to substrate by a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-FEM1C with trapped UBE2R2~donor UB-Sil1 peptide

PDB-8r5h: 
Ubiquitin ligation to neosubstrate by a cullin-RING E3 ligase & Cdc34: NEDD8-CUL2-RBX1-ELOB/C-VHL-MZ1 with trapped UBE2R2~donor UB-BRD4 BD2

EMDB-16433: 
Cryo-EM structure of NADH bound SLA dehydrogenase RlGabD from Rhizobium leguminosarum bv. trifolii SRD1565

PDB-8c54: 
Cryo-EM structure of NADH bound SLA dehydrogenase RlGabD from Rhizobium leguminosarum bv. trifolii SRD1565

EMDB-16144: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the macrocyclic peptide S1B3inL1

PDB-8bon: 
Structure of the SARS-CoV-2 spike glycoprotein in complex with the macrocyclic peptide S1B3inL1

EMDB-40422: 
Cryo-EM Structure of RyR1

EMDB-40423: 
Cryo-EM Structure of RyR1 + ATP-gamma-S

EMDB-40424: 
Cryo-EM Structure of RyR1 + ADP

EMDB-40425: 
Cryo-EM Structure of RyR1 + AMP

EMDB-40426: 
Cryo-EM Structure of RyR1 + Adenosine

EMDB-40427: 
Cryo-EM Structure of RyR1 + Adenine

EMDB-40428: 
Cryo-EM Structure of RyR1 + cAMP

EMDB-40429: 
Cryo-EM Structure of RyR1 (Local Refinement of TMD)

EMDB-40430: 
Cryo-EM Structure of RyR1 + ATP-gamma-S (Local Refinement of TMD)

EMDB-40431: 
Cryo-EM Structure of RyR1 + ADP (Local Refinement of TMD)

EMDB-40432: 
Cryo-EM Structure of RyR1 + AMP (Local Refinement of TMD)

EMDB-40433: 
Cryo-EM Structure of RyR1 + Adenosine (Local Refinement of TMD)

EMDB-40434: 
Cryo-EM Structure of RyR1 + Adenine (Local Refinement of TMD)

EMDB-40435: 
Cryo-EM Structure of RyR1 + cAMP (Local Refinement of TMD)

PDB-8sen: 
Cryo-EM Structure of RyR1

PDB-8seo: 
Cryo-EM Structure of RyR1 + ATP-gamma-S

PDB-8sep: 
Cryo-EM Structure of RyR1 + ADP

PDB-8seq: 
Cryo-EM Structure of RyR1 + AMP

PDB-8ser: 
Cryo-EM Structure of RyR1 + Adenosine

PDB-8ses: 
Cryo-EM Structure of RyR1 + Adenine

PDB-8set: 
Cryo-EM Structure of RyR1 + cAMP

PDB-8seu: 
Cryo-EM Structure of RyR1 (Local Refinement of TMD)

PDB-8sev: 
Cryo-EM Structure of RyR1 + ATP-gamma-S (Local Refinement of TMD)

PDB-8sew: 
Cryo-EM Structure of RyR1 + ADP (Local Refinement of TMD)

PDB-8sex: 
Cryo-EM Structure of RyR1 + AMP (Local Refinement of TMD)

PDB-8sey: 
Cryo-EM Structure of RyR1 + Adenosine (Local Refinement of TMD)

PDB-8sez: 
Cryo-EM Structure of RyR1 + Adenine (Local Refinement of TMD)

PDB-8sf0: 
Cryo-EM Structure of RyR1 + cAMP (Local Refinement of TMD)
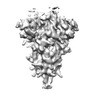
EMDB-16403: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C3 symmetry
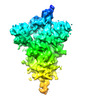
EMDB-16404: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry

EMDB-16405: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E) in C3 symmetry

EMDB-16406: 
Subtomogram averaging of a coronavirus spike in situ (ChAdOx1 19E) in C1 symmetry

EMDB-16697: 
Subtomogram averaging of a coronavirus spike (ChAdOx1 19E6) in C1 symmetry after 3D classification
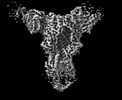
EMDB-26983: 
1F8 mAb in complex with the computationally optimized broadly reactive H1 influenza hemagglutinin P1

PDB-8ct6: 
1F8 mAb in complex with the computationally optimized broadly reactive H1 influenza hemagglutinin P1

EMDB-27973: 
Erwinia amylovora 70S Ribosome

EMDB-27993: 
Erwinia amlyavora 50S ribosome

EMDB-28003: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Vertex

EMDB-28004: 
Erwinia phage vB_EamM_RAY (RAY) Capsid Collar
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
