-検索条件
-検索結果
検索 (著者・登録者: shi & yf)の結果全43件を表示しています
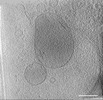
EMDB-39012: 
Representative tomogram of primary glioblastoma stem cell with circular inter-mitochondrial junctions.

EMDB-39015: 
Representative tomogram of microglia cell with nanotunnel-like structures resembling mitochondrial fission.
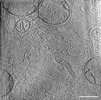
EMDB-39019: 
Representative tomogram of glioblastoma cell with nanotunnel-like structure and inter-mitochondrial junction.

EMDB-39021: 
Representative tomogram of normal human astrocyte with nanotunnel-like structure which is an extension of the mitochondrial outer membrane.

EMDB-39023: 
Representative tomogram of primary glioblastoma differentiated cell with parallel inter-mitochondrial junction.

EMDB-39024: 
Representative tomogram of primary glioblastoma stem cell with clustered mitochondria bearing various long-short axis ratios.

EMDB-29943: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29944: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29945: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29946: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 3 Coupled to G Protein

EMDB-29935: 
Cryo-EM Structure of the Prostaglandin E Receptor EP4 Coupled to G Protein

EMDB-29940: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-35063: 
The complex structure of Omicron BA.4 RBD with BD604, S309, and S304

EMDB-29016: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane
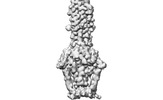
EMDB-29017: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-29018: 
Cryo-EM structure of SARS-CoV-2 postfusion spike in membrane

EMDB-34731: 
The structure of MPXV polymerase holoenzyme in replicating state

EMDB-25648: 
CryoEM structure of the adenosine 2A receptor-BRIL/Anti BRIL Fab complex with ZM241385

EMDB-32944: 
The complex structure of Omicron BA.1 RBD with BD604, S309,and S304

EMDB-32856: 
Cryo-EM structure of SARS-CoV-2 Omicron Spike protein with human ACE2 receptor, C2 state

EMDB-32857: 
Cryo-EM structure of SARS-CoV-2 Omicron Spike protein with human ACE2 receptor, C3 state

EMDB-32558: 
Cryo-EM structure of SARS-CoV-2 Omicron spike protein with ACE2, C1 state

EMDB-32559: 
Cryo-EM structure of Omicron S-ACE2, C2 state

EMDB-32560: 
Cryo-EM structure of SARS-CoV-2 Omicron spike protein with human ACE2 (focus refinement on RBD-1/ACE2)

EMDB-32428: 
RBD-1 of SARS-CoV-2 Beta spike in complex with S5D2 Fab

EMDB-32430: 
SARS-CoV-2 Beta spike in complex with one S5D2 Fab

EMDB-32431: 
SARS-CoV-2 Beta spike in complex with two S5D2 Fabs

EMDB-32433: 
SARS-CoV-2 Beta spike in complex with three S5D2 Fabs

EMDB-32434: 
SARS-CoV-2 Beta spike SD1 in complex with S3H3 Fab

EMDB-32435: 
SARS-CoV-2 Beta spike in complex with three S3H3 Fabs

EMDB-32437: 
SARS-CoV-2 Beta spike in complex with two S3H3 Fabs

EMDB-32170: 
SARS-CoV-2 Beta variant spike protein in transition state

EMDB-31542: 
SARS-COV-2 Spike RBDMACSp6 binding to hACE2

EMDB-31543: 
SARS-COV-2 Spike RBDMACSp25 binding to hACE2

EMDB-31544: 
SARS-COV-2 Spike RBDMACSp36 binding to hACE2

EMDB-31546: 
SARS-COV-2 Spike RBDMACSp36 binding to mACE2

EMDB-30702: 
S-2H2-F3a structure, two RBDs are up and one RBD is down, each RBD binds with a 2H2 Fab.

EMDB-30703: 
S-2H2-F1 structure, one RBD is up and two RBDs are down, only up RBD binds with a 2H2 Fab

EMDB-30704: 
S-2H2-F2 structure, two RBDs are up and one RBD is down, each up RBD binds with a 2H2 Fab.

EMDB-30705: 
S-2H2-F3b structure, three RBDs are up and each RBD binds with a 2H2 Fab.

EMDB-3125: 
Structural basis for specific recognition of single-stranded RNA by toll-like receptor 13

EMDB-3026: 
Electron cryo-microscopy of porcine Factor VIII bound to lipid nanotubes and single particle tomography reconstruction

EMDB-3027: 
Electron cryo-microscopy of porcine Factor VIII bound to lipid nanotubes and helical reconstruction
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
