-検索条件
-検索結果
検索 (著者・登録者: rey & f)の結果2,188件中、1から50件目までを表示しています

EMDB-42603: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/hexamer)

EMDB-42625: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC804515)

EMDB-42626: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/up)

EMDB-42627: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/down)

EMDB-44748: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/dodecamer)

PDB-8uv2: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/hexamer)

PDB-8uvo: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC804515)

PDB-8uvp: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/up)

PDB-8uvq: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/down)

PDB-9boq: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/dodecamer)
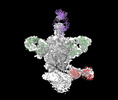
EMDB-44246: 
Cryo-EM structure of HIV-1 JRFL v6 Env in complex with vaccine-elicited, Membrane Proximal External Region (MPER) directed antibody DH1317.4.

EMDB-43128: 
Structure of the human dopamine transporter in complex with beta-CFT, MRS7292 and divalent zinc

PDB-8vby: 
Structure of the human dopamine transporter in complex with beta-CFT, MRS7292 and divalent zinc

EMDB-45430: 
Human Mitochondrial LONP1 Degrading Casein, ATP-bound closed form

EMDB-45433: 
Human Mitochondrial LONP1 Stall State + casein

EMDB-45434: 
LONP1 stall state bound to substrate and 4 ADPs

PDB-9cc0: 
Human Mitochondrial LONP1 Degrading Casein, ATP-bound closed form

PDB-9cc3: 
Human Mitochondrial LONP1 Stall State + casein

EMDB-43139: 
SARS-CoV-2 Spike S2 bound to Fab 54043-5

EMDB-18402: 
cryo-EM structure of apo-TcdB

EMDB-18403: 
cryo-EM map of apo Clostridioides difficile toxin B

EMDB-18409: 
cryo-EM structure of TcdB-FZD7

EMDB-18410: 
cryo-EM structure of TcdB-FZD7

EMDB-18411: 
cryo-EM structure of TcdB-FZD7

EMDB-50860: 
Cryo EM map of the type 2A polymorph of alpha-synuclein at pH 7.0.

EMDB-50888: 
Cryo EM structure of the type 3B polymorph of alpha-synuclein at low pH.

PDB-9fyp: 
Cryo EM structure of the type 3B polymorph of alpha-synuclein at low pH.
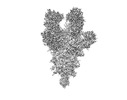
EMDB-18399: 
SARS-CoV-2 Spike in complex with the neutralizing antibody Cv2.3194

EMDB-43592: 
PDI-containing spoke of a hexagonal wireframe DNA origami

EMDB-18313: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

EMDB-18314: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

EMDB-18315: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-18317: 
Retron-Eco1 filament (2 segments)

EMDB-19792: 
Retron-Eco1 -1 turn mutant filament with ADP-ribosylated Effector (Consensus refinement)

EMDB-19793: 
Retron-Eco1 filament with ADP-ribosylated Effector (Consensus refinement)

PDB-8qbk: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

PDB-8qbl: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

PDB-8qbm: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-42150: 
Human Mitochondrial DNA Polymerase Gamma Binary Complex

PDB-8udl: 
Human Mitochondrial DNA Polymerase Gamma Binary Complex

EMDB-42676: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

EMDB-42999: 
5HT2AR-miniGq heterotrimer in complex with a novel agonist obtained from large scale docking

PDB-8uwl: 
5-HT2AR bound to Lisuride in complex with a mini-Gq protein and an active-state stabilizing single-chain variable fragment (scFv16) obtained by cryo-electron microscopy (cryoEM)

PDB-8v6u: 
5HT2AR-miniGq heterotrimer in complex with a novel agonist obtained from large scale docking

EMDB-17693: 
Cryo EM structure of the type 3C polymorph of alpha-synuclein at low pH.

EMDB-17714: 
Cryo EM structure of the type 3D polymorph of alpha-synuclein E46K mutant at low pH.

EMDB-17723: 
Cryo EM structure of the type 1m polymorph of alpha-synuclein

EMDB-17726: 
Cryo EM structure of the type 5A polymorph of alpha-synuclein.

EMDB-50077: 
Cryo EM map of the type 2B polymorph of alpha-synuclein at pH 7.0.

PDB-8pix: 
Cryo EM structure of the type 3C polymorph of alpha-synuclein at low pH.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
