-検索条件
-検索結果
検索 (著者・登録者: o & donnell & j)の結果62件中、1から50件目までを表示しています
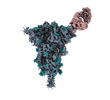
EMDB-40812: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 1

EMDB-40813: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 2

EMDB-40814: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

PDB-8swh: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

EMDB-42399: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp and DNA

EMDB-42402: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp

PDB-8unf: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp and DNA

PDB-8unh: 
Cryo-EM structure of T4 Bacteriophage Clamp Loader with Sliding Clamp
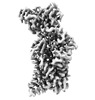
EMDB-26160: 
Structure of human serotonin transporter bound to small molecule '8090 in lipid nanodisc and NaCl

PDB-7txt: 
Structure of human serotonin transporter bound to small molecule '8090 in lipid nanodisc and NaCl

EMDB-27488: 
Cryo-EM structure of cystinosin in a cytosol-open state

EMDB-27489: 
Cryo-EM structure of cystinosin in a lumen-open state

EMDB-27490: 
Cryo-EM structure of cystine-bound cystinosin in a lumen-open state

EMDB-27492: 
Cryo-EM structure of cystinosin N288K mutant in a cytosol-open state at pH5.0

EMDB-27493: 
Cryo-EM structure of cystinosin N288K mutant in a cytosol-open state at pH7.5

PDB-8dke: 
Cryo-EM structure of cystinosin in a cytosol-open state

PDB-8dki: 
Cryo-EM structure of cystinosin in a lumen-open state

PDB-8dkm: 
Cryo-EM structure of cystine-bound cystinosin in a lumen-open state

PDB-8dkw: 
Cryo-EM structure of cystinosin N288K mutant in a cytosol-open state at pH5.0

PDB-8dkx: 
Cryo-EM structure of cystinosin N288K mutant in a cytosol-open state at pH7.5

EMDB-24310: 
The structure of human ABCG5-WT/ABCG8-I419E

EMDB-24311: 
The structure of human ABCG5-I529W/ABCG8-WT

EMDB-24312: 
The structure of human ABCG5/ABCG8 purified from yeast

EMDB-24313: 
The structure of human ABCG5/ABCG8 purified from mammalian cells

EMDB-24314: 
The structure of human ABCG5/ABCG8 supplemented with cholesterol

EMDB-24315: 
The structure of human ABCG1

EMDB-24316: 
The structure of human ABCG1 E242Q with cholesterol

EMDB-24317: 
The structure of human ABCG1 E242Q complexed with ATP

PDB-7r87: 
The structure of human ABCG5-WT/ABCG8-I419E

PDB-7r88: 
The structure of human ABCG5-I529W/ABCG8-WT

PDB-7r89: 
The structure of human ABCG5/ABCG8 purified from yeast

PDB-7r8a: 
The structure of human ABCG5/ABCG8 purified from mammalian cells

PDB-7r8b: 
The structure of human ABCG5/ABCG8 supplemented with cholesterol

PDB-7r8c: 
The structure of human ABCG1

PDB-7r8d: 
The structure of human ABCG1 E242Q with cholesterol

PDB-7r8e: 
The structure of human ABCG1 E242Q complexed with ATP

EMDB-12530: 
Subtomogram average of ChAdOx1 nCoV-19/AZD1222 derived SARS-CoV-2 spike glycoprotein

PDB-6z3w: 
Human ER membrane protein complex

EMDB-10913: 
Structure of the phage STP1 host adhesion device (baseplate) by negative staining

EMDB-11058: 
Human ER Membrane protein Complex (EMC)

EMDB-8519: 
Structure of Eukaryotic CMG Helicase at a Replication Fork and Implications

PDB-5u8t: 
Structure of Eukaryotic CMG Helicase at a Replication Fork and Implications

EMDB-8518: 
Structure of eukaryotic CMG helicase at a replication fork

EMDB-8520: 
Structure of Eukaryotic CMG Helicase at a Replication Fork and Implications to Replisome Architecture and Origin Initiation

PDB-5u8s: 
Structure of eukaryotic CMG helicase at a replication fork

EMDB-6534: 
Structure of the eukaryotic replicative CMG helicase and pumpjack motion

EMDB-6535: 
Structure of the eukaryotic replicative CMG helicase and pumpjack motion

EMDB-6536: 
Structure of the eukaryotic replicative CMG helicase and pumpjack motion

PDB-3jc5: 
Structure of the eukaryotic replicative CMG helicase and pumpjack motion

PDB-3jc6: 
Structure of the eukaryotic replicative CMG helicase and pumpjack motion
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
