-検索条件
-検索結果
検索 (著者・登録者: ma & xm)の結果59件中、1から50件目までを表示しています

EMDB-35042: 
Cryo-EM structure of the the 2-oxoglutarate dehydrogenase (E1) with TCAIM complex

EMDB-17766: 
CryoEM structure of Nal1 protein, allele SPIKE, from Oryza sativa japonica group
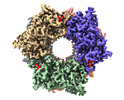
EMDB-17768: 
CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar
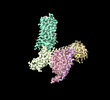
EMDB-35297: 
Structural insights into human brain gut peptide cholecystokinin receptors

EMDB-15417: 
human MutSalpha (MSH2/MSH6) binding to DNA with a GT mismatch

EMDB-15519: 
human MutSalpha (MSH2/MSH6) on DNA containing a GT mismatch in the presence of ADP

PDB-8ag6: 
human MutSalpha (MSH2/MSH6) binding to DNA with a GT mismatch

EMDB-15518: 
human MutSalpha mutant (MSH2-V63E/MSH6) on DNA containing a GT mismatch

EMDB-26030: 
Structure of G6PD-WT tetramer with no symmetry imposed

EMDB-26031: 
Structure of G6PD-WT dimer with no symmetry applied

EMDB-26428: 
Structure of G6PD-D200N tetramer bound to NADP+ and G6P with no symmetry applied

EMDB-26442: 
Structure of G6PD-D200N tetramer bound to NADP+ with no symmetry applied

PDB-7toe: 
Structure of G6PD-WT tetramer with no symmetry imposed

PDB-7tof: 
Structure of G6PD-WT dimer with no symmetry applied

PDB-7ual: 
Structure of G6PD-D200N tetramer bound to NADP+ and G6P with no symmetry applied

PDB-7uc2: 
Structure of G6PD-D200N tetramer bound to NADP+ with no symmetry applied

EMDB-33359: 
Structural insights into human brain gut peptide cholecystokinin receptors

EMDB-33360: 
Structural insights into human brain gut peptide cholecystokinin receptors

EMDB-33361: 
Structural insights into human brain gut peptide cholecystokinin receptors

EMDB-25224: 
Structure of G6PD-WT dimer

EMDB-25225: 
structure of G6PD-WT tetramer

EMDB-25226: 
Structure of G6PD-D200N tetramer bound to NADP+

EMDB-25227: 
Structure of G6PD-D200N tetramer bound to NADP+ and G6P

PDB-7snf: 
Structure of G6PD-WT dimer

PDB-7sng: 
structure of G6PD-WT tetramer

PDB-7snh: 
Structure of G6PD-D200N tetramer bound to NADP+

PDB-7sni: 
Structure of G6PD-D200N tetramer bound to NADP+ and G6P

EMDB-32485: 
Cryo-electron microscopic structure of the 2-oxoglutarate dehydrogenase (E1) component of the human alpha-ketoglutarate (2-oxoglutarate) dehydrogenase complex

EMDB-33331: 
Cyo-EM model for native cystathionine beta-synthase of Mycobacterium tuberculosis.
EMDB-33348: 
Cryo-EM map of cystathionine beta-synthase of Mycobacterium tuberculosis in the presence of S-adenosylmethionine.
EMDB-33363: 
Cryo-EM map of cystathionine beta-synthase of Mycobacterium tuberculosis in the presence of S-adenosylmethionine and serine.

PDB-7xnz: 
Native cystathionine beta-synthase of Mycobacterium tuberculosis.

PDB-7xoh: 
Cystathionine beta-synthase of Mycobacterium tuberculosis in the presence of S-adenosylmethionine.

PDB-7xoy: 
Cystathionine beta-synthase of Mycobacterium tuberculosis in the presence of S-adenosylmethionine and serine.

EMDB-31246: 
Cryo-electron tomogram of phycobilisome-photosystem II supercomplex on the thylakoid

EMDB-31241: 
Structure of the PBS-PSII supercomplex from red algae

EMDB-31242: 
Structure of the double phycobilisome-photosystem II supercomplex from red alga

EMDB-31243: 
Cryo-electron tomography of phycobilisome-photosystem II supercomplex on the thylakoid

EMDB-31244: 
Cryo-electron tomogram of phycobilisome-photosystem II supercomplex on the thylakoid

EMDB-31245: 
Cryo-electron tomogram of phycobilisome-photosystem II supercomplex on the thylakoid

EMDB-31247: 
Cryo-electron tomogram of phycobilisome-photosystem II supercomplex on the thylakoid

EMDB-30650: 
Cryo-EM structure of the Ams1 and Nbr1 complex

EMDB-30652: 
Cryo-EM structure of the Ape4 and Nbr1 complex

EMDB-11791: 
MutS in Scanning state

EMDB-11792: 
MutS in mismatch bound state

EMDB-11793: 
MutS in Intermediate state

EMDB-11794: 
MutS-MutL in clamp state

EMDB-11795: 
MutS-MutL in clamp state (kinked clamp domain)

PDB-7ai5: 
MutS in Scanning state

PDB-7ai6: 
MutS in mismatch bound state
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
