-検索条件
-検索結果
検索 (著者・登録者: lazaro & m)の結果全46件を表示しています

EMDB-42804: 
Structure of nucleotide-free Pediculus humanus (Ph) PINK1 dimer

EMDB-42806: 
Structure of AMP-PNP-bound Pediculus humanus (Ph) PINK1 dimer

EMDB-42807: 
Structure of ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dimer

PDB-8uyf: 
Structure of nucleotide-free Pediculus humanus (Ph) PINK1 dimer

PDB-8uyh: 
Structure of AMP-PNP-bound Pediculus humanus (Ph) PINK1 dimer

PDB-8uyi: 
Structure of ADP-bound and phosphorylated Pediculus humanus (Ph) PINK1 dimer
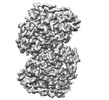
EMDB-15036: 
Cryo-EM structure of "CT-CT dimer" of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz6: 
Cryo-EM structure of "CT-CT dimer" of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-13682: 
In situ subtomogram average of autophagosome-associated protein filament

EMDB-15390: 
Subtomogram average of empty poliovirus particles

EMDB-15391: 
Subtomogram average of RNA-loaded poliovirus

EMDB-15392: 
Subtomogram average of membrane-tethered poliovirus

EMDB-15028: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15029: 
Cryo-EM structure of "CT oxa" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA
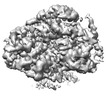
EMDB-15030: 
Cryo-EM structure of "CT empty" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA
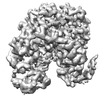
EMDB-15031: 
Cryo-EM structure of "CT react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15032: 
Cryo-EM structure of "CT pyr" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15033: 
Cryo-EM structure of "BC react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15034: 
Cryo-EM structure of "BC closed" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15035: 
Cryo-EM structure of "BC open" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

EMDB-15037: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA and cyclic di-AMP

PDB-7zyy: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zyz: 
Cryo-EM structure of "CT oxa" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz0: 
Cryo-EM structure of "CT empty" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz1: 
Cryo-EM structure of "CT react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz2: 
Cryo-EM structure of "CT pyr" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz3: 
Cryo-EM structure of "BC react" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz4: 
Cryo-EM structure of "BC closed" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz5: 
Cryo-EM structure of "BC open" conformation of Lactococcus lactis pyruvate carboxylase with acetyl-CoA

PDB-7zz8: 
Cryo-EM structure of Lactococcus lactis pyruvate carboxylase with acetyl-CoA and cyclic di-AMP

EMDB-24144: 
ApoL3, A human apolipoprotein L

EMDB-11606: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (open conformation)

EMDB-11612: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (close conformation)

EMDB-11613: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (monomer)

PDB-7a1d: 
Cryo-EM map of the large glutamate dehydrogenase composed of 180 kDa subunits from Mycobacterium smegmatis (open conformation)

EMDB-23265: 
Computationally designed icosahedral antibody nanocage with Fc i52.3+Fc

EMDB-23266: 
Computationally designed octahedral antibody nanocage with Fc o42.1+Fc

EMDB-23120: 
3D reconstruction of an autophagy tethering factor

EMDB-20645: 
MicroED structure of a FIB-milled CypA Crystal

PDB-6u5g: 
MicroED structure of a FIB-milled CypA Crystal

EMDB-5944: 
Pyruvate Carboxylase tetramer in symmetric architecture

EMDB-5945: 
Pyruvate Carboxylase tetramer in asymmetric architecture

EMDB-2048: 
Cryo-EM structure of the UPF-EJC complex

EMDB-1551: 
Pretranslocation ribosome. Total population.

EMDB-1553: 
Pretranslocation ribosome. Group after ML3D classification. Racheted ribosome with tRNAs in hybrid positions.

EMDB-1554: 
Pretranslocation ribosome. Group after ML3D classification. Non-racheted and classical tRNA positions.
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
