-検索条件
-検索結果
検索 (著者・登録者: jung & y)の結果295件中、1から50件目までを表示しています

EMDB-44148: 
The cryo-EM structure of the H2A.Z-H3.3 double-variant nucleosome

PDB-9b3p: 
The cryo-EM structure of the H2A.Z-H3.3 double-variant nucleosome

EMDB-19798: 
human PLD3 homodimer structure

PDB-8s86: 
human PLD3 homodimer structure

EMDB-36800: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

EMDB-36801: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrA octamer

EMDB-36802: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrB dimer

EMDB-36803: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

EMDB-36804: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

EMDB-38477: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

EMDB-38478: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry

PDB-8k1s: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state

PDB-8k1t: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2

PDB-8k1u: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA

PDB-8xmh: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis

PDB-8xmi: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry

EMDB-17630: 
ABCB1 L335C mutant (mABCB1) in the inward facing state bound to AAC

PDB-8pee: 
ABCB1 L335C mutant (mABCB1) in the inward facing state bound to AAC
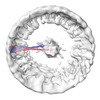
EMDB-18698: 
Structural insights into the activation mechanism of antimicrobial GBP1: Polymeric assembly of GBP1

EMDB-18806: 
Structural insights into the activation mechanism of antimicrobial GBP1: Membrane-bound GBP1 oligomer

PDB-8r1a: 
Model of the membrane-bound GBP1 oligomer

EMDB-35010: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

EMDB-35770: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Consensus map)

EMDB-35772: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Receptor-focused map)

EMDB-35773: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (G protein-focused map)
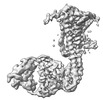
EMDB-35971: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL
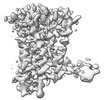
EMDB-37336: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

PDB-8hti: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

PDB-8j46: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL

PDB-8w77: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

EMDB-41280: 
Non-targeted transpososome from ShCAST

EMDB-35904: 
AtSLAC1 8D mutant in closed state

EMDB-35920: 
AtSLAC1 in open state

PDB-8j0j: 
AtSLAC1 8D mutant in closed state

PDB-8j1e: 
AtSLAC1 in open state

EMDB-34303: 
AtSLAC1 6D mutant in closed state

EMDB-34304: 
AtSLAC1 6D mutant in open state

PDB-8gw6: 
AtSLAC1 6D mutant in closed state

PDB-8gw7: 
AtSLAC1 6D mutant in open state

EMDB-29013: 
96nm doublet microtubule repeat from wild type mouse sperm

EMDB-28606: 
96nm doublet microtubule repeat from LRRC23-KO mouse sperm

EMDB-15687: 
The ABCB1 L335C mutant (mABCB1) in the Apo state

PDB-8avy: 
The ABCB1 L335C mutant (mABCB1) in the Apo state

EMDB-27781: 
Cryo-EM structure of 227 Fab in complex with (NPNA)8 peptide

EMDB-27784: 
Cryo-EM structure of 239 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27785: 
Cryo-EM structure of 311 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27786: 
Cryo-EM structure of 334 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27787: 
Cryo-EM structure of 337 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27788: 
Cryo-EM structure of 356 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)

EMDB-27789: 
Cryo-EM structure of 364 Fab in complex with recombinant shortened Plasmodium falciparum circumsporozoite protein (rsCSP)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
