-検索条件
-検索結果
検索 (著者・登録者: jones & ja)の結果172件中、1から50件目までを表示しています

EMDB-17295: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '3 up' RBD conformation

PDB-8oyt: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '3 up' RBD conformation

EMDB-17296: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

PDB-8oyu: 
Stabilised BA.1 SARS-CoV-2 spike with H6 nanobodies in '2 up 1 down' RBD conformation

EMDB-41904: 
Klebsiella pneumoniae encapsulin-associated DyP peroxidase

EMDB-41905: 
Klebsiella pneumoniae DyP peroxidase-loaded encapsulin shell

EMDB-41906: 
Klebsiella pneumoniae SUMO-loaded encapsulin shell

EMDB-16816: 
Structure of TolQR complex from E.coli

PDB-8odt: 
Structure of TolQR complex from E.coli

EMDB-17586: 
E. coli RNA polymerase elongation complex stalled at thymine dimer lesion

PDB-8pbl: 
E. coli RNA polymerase elongation complex stalled at thymine dimer lesion

EMDB-16052: 
Cryo-EM structure of stalled rabbit 80S ribosomes in complex with human CCR4-NOT and CNOT4

EMDB-29857: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

PDB-8g8w: 
Molecular mechanism of nucleotide inhibition of human uncoupling protein 1

EMDB-29396: 
Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP
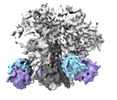
EMDB-29836: 
vFP52.02 Fab in complex with BG505 DS-SOSIP Env trimer

EMDB-29880: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 1)

EMDB-29881: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 2)

EMDB-29882: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 3)

EMDB-29905: 
vFP48.02 Fab in complex with BG505 DS-SOSIP Env trimer

PDB-8fr6: 
Antibody vFP53.02 in complex with HIV-1 envelope trimer BG505 DS-SOSIP

PDB-8g85: 
vFP52.02 Fab in complex with BG505 DS-SOSIP Env trimer

PDB-8g9w: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 1)

PDB-8g9x: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 2)

PDB-8g9y: 
Cryo-EM structure of vFP49.02 Fab in complex with HIV-1 Env BG505 DS-SOSIP.664 (conformation 3)

PDB-8gas: 
vFP48.02 Fab in complex with BG505 DS-SOSIP Env trimer

EMDB-27558: 
Acidipropionibacterium acidipropionici encapsulin in a closed state at pH 7.5

EMDB-27560: 
Acidipropionibacterium acidipropionici encapsulin in a closed state at pH 3.0

EMDB-27573: 
Acidipropionibacterium acidipropionici encapsulin in an open state at pH 7.5

EMDB-27385: 
Oligomeric C9 in complex with aE11 Fab

EMDB-28863: 
PolyC9 in complex with aE11 Fab

PDB-8de6: 
Oligomeric C9 in complex with aE11 Fab
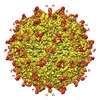
EMDB-25606: 
Zika Virus particle bound with IgM antibody DH1017 Fab fragment

EMDB-26419: 
Human pro-meprin alpha (zymogen state)[subparticle localised reconstruction]

EMDB-26420: 
Human pro-meprin alpha (zymogen state)[Focused classification and refinement]

EMDB-26421: 
Meprin alpha helix (activated) - full C1 consensus reconstruction

EMDB-26422: 
Human meprin alpha (active state)[subparticle localised reconstruction]

EMDB-26423: 
Human meprin alpha inhibitory complex with compound 10d (N~3~,N~3~-bis[(2H-1,3-benzodioxol-5-yl)methyl]-N-hydroxy-beta-alaninamide)

EMDB-26424: 
Meprin alpha helix in complex with fetuin-B [consensus C1 reconstruction]

EMDB-26426: 
Meprin alpha helix in complex with fetuin-B [subparticle localised reconstruction, masked focused refinement]

EMDB-27689: 
Sub-tomogram average of pro-meprin alpha supercoiled filament

PDB-7uab: 
Human pro-meprin alpha (zymogen state)

PDB-7uac: 
Human pro-meprin alpha (zymogen state)

PDB-7uae: 
Human meprin alpha (active state)

PDB-7uaf: 
Human meprin alpha inhibitory complex with compound 10d (N~3~,N~3~-bis[(2H-1,3-benzodioxol-5-yl)methyl]-N-hydroxy-beta-alaninamide)

PDB-7uai: 
Meprin alpha helix in complex with fetuin-B

EMDB-25524: 
Reconstruction of full-length Prex-1 (PtdIns(3,4,5)P3-dependent Rac Exchanger 1)

EMDB-25525: 
Localised reconstruction of the N-terminal half of P-Rex1 (PI(3,4,5)P3-dependent Rac Exchanger 1)

EMDB-25526: 
Localised reconstruction of the C-terminal half of P-Rex 1 (PI(3,4,5)P3-dependent Rac Exchanger 1)

PDB-7syf: 
Reconstruction of full-length Prex-1 (PtdIns(3,4,5)P3-dependent Rac Exchanger 1)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
