-検索条件
-検索結果
検索 (著者・登録者: jeffrey & pd)の結果98件中、1から50件目までを表示しています
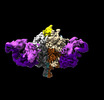
EMDB-41048: 
Lassa GPC Trimer in complex with Fab 8.11G and nanobody D5

EMDB-29447: 
Sec39:Use1:Sec20:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex #2

EMDB-29292: 
HIV-1 envelope trimer bound to one CD4 molecule on membranes

EMDB-29293: 
HIV-1 envelope trimer bound to two CD4 molecules on membranes

EMDB-29294: 
HIV-1 envelope trimer bound to three CD4 molecules on membranes

EMDB-29295: 
Consensus structure of HIV-1 envelope trimer bound to CD4 receptors on membranes

EMDB-41045: 
Unliganded HIV-1 Env on virion

EMDB-28760: 
Dsl1:Sec39 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28762: 
Dsl1:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28768: 
Sec39:Use1:Sec20:Tip20 Local Refinement of the Dsl1:Qb:Qc Complex

EMDB-28774: 
Consensus Map of the Dsl1:Qb:Qc Complex

EMDB-28204: 
CryoEM structure of the Dsl1 complex bound to SNAREs Sec20 and Use1

EMDB-41302: 
Lassa GPC trimer in complex with Fab GP23

EMDB-26859: 
Ligand-free Lassa GPC Trimer with C3 Symmetry

EMDB-26740: 
Ligand-free Lassa GPC Trimer with C1 Symmetry

EMDB-28092: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-093

EMDB-28090: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-040

EMDB-28091: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-045
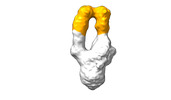
EMDB-28093: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-156
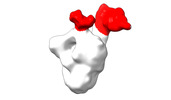
EMDB-28094: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-234

EMDB-28095: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-260

EMDB-28096: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-279

EMDB-28097: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-290
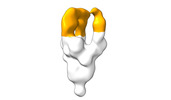
EMDB-28098: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-294

EMDB-28099: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-295

EMDB-28100: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-299

EMDB-28102: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-334
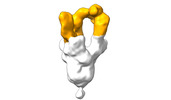
EMDB-28103: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-360

EMDB-28104: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-361

EMDB-28105: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-362

EMDB-28106: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-368

EMDB-28168: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-292

EMDB-28169: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-333
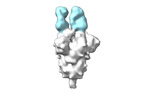
EMDB-28170: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-355

EMDB-28171: 
Negative stain EM map of SARS-CoV-2 Spike in complex with CoVIC-371

EMDB-26044: 
H10ssF: ferritin-based nanoparticle displaying H10 hemagglutinin stabilized stem epitopes

EMDB-27735: 
Cryo-EM structure of SIVmac239 SOS-2P Env trimer in complex with human bNAb PGT145

EMDB-27718: 
SIV E660.CR54 SOS-2P Env Trimer with ITS92.02

EMDB-25062: 
In-situ structure of SIV trimer

EMDB-25063: 
in situ SIVmac239-Env trimers on the surface of AT-2-inactivated virions

EMDB-25064: 
in situ SIVmac239-Env trimers on the surface of AT-2-inactivated virions

EMDB-25065: 
In-situ structure of SIVmac239-Env trimers with ITS90.03 associated
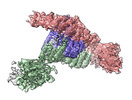
EMDB-27442: 
Cryo-EM structure of a HOPS core complex containing Vps33, Vps16, and Vps18

EMDB-27631: 
SIV mac239 SOS-2P K169T Env trimer with bNAb PGT145 and jacalin
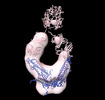
EMDB-26217: 
Negative stain EM map of COVA1-07 mAb bound to the S2 domain of SARS-CoV-2 S
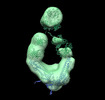
EMDB-26218: 
Negative stain EM map of COVA2-14 mAb bound to the S2 domain of SARS-CoV-2 S

EMDB-26219: 
Negative stain EM map of COVA2-18 mAb bound to the S2 domain of SARS-CoV-2 S
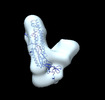
EMDB-26220: 
Negative stain EM map of the S2 domain of SARS-CoV-2 S

EMDB-23098: 
Cryo-EM structure of the VRC316 clinical trial, vaccine-elicited, human antibody 316-310-1B11 in complex with an H2 CAN05 HA trimer

EMDB-23816: 
Cryo-EM structure of the VRC310 clinical trial, vaccine-elicited, human antibody 310-030-1D06 Fab in complex with an H1 NC99 HA trimer
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
