-検索条件
-検索結果
検索 (著者・登録者: fang & w)の結果1,772件中、1から50件目までを表示しています
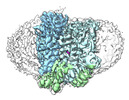
EMDB-38966: 
Cryo-EM structure of human urate transporter GLUT9 bound to substrate urate
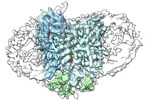
EMDB-38968: 
Cryo-EM structure of human urate transporter GLUT9 bound to inhibitor apigenin

PDB-8y65: 
Cryo-EM structure of human urate transporter GLUT9 bound to substrate urate

PDB-8y66: 
Cryo-EM structure of human urate transporter GLUT9 bound to inhibitor apigenin

EMDB-37249: 
Cryo-EM structure of EBV gH/gL-gp42 in complex with fab 2C1

PDB-8khr: 
Cryo-EM structure of EBV gH/gL-gp42 in complex with fab 2C1

EMDB-38216: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

PDB-8xbf: 
Cryo-EM structure of SARS-CoV-2 S-BQ.1 in complex with antibody O5C2

EMDB-37754: 
Fe-O nanocluster of form-IX in the 4-fold channel of Ureaplasma diversum ferritin

EMDB-37755: 
Fe-O nanocluster of form-VIII in the 4-fold channel of Ureaplasma diversum ferritin

EMDB-37757: 
Fe-O nanocluster of form-X in the 4-fold channel of Ureaplasma diversum ferritin

EMDB-37758: 
Fe-O nanocluster of form-XI in the 4-fold channel of Ureaplasma diversum ferritin

EMDB-37759: 
Fe-O nanocluster of form-XII in the 4-fold channel of Ureaplasma diversum ferritin

PDB-8wqu: 
Fe-O nanocluster of form-IX in the 4-fold channel of Ureaplasma diversum ferritin

PDB-8wqv: 
Fe-O nanocluster of form-VIII in the 4-fold channel of Ureaplasma diversum ferritin

PDB-8wqx: 
Fe-O nanocluster of form-X in the 4-fold channel of Ureaplasma diversum ferritin

PDB-8wqy: 
Fe-O nanocluster of form-XI in the 4-fold channel of Ureaplasma diversum ferritin

PDB-8wr0: 
Fe-O nanocluster of form-XII in the 4-fold channel of Ureaplasma diversum ferritin

EMDB-36907: 
Cryo-EM structure of the RC-LH core comples from Halorhodospira halochloris

PDB-8k5o: 
Cryo-EM structure of the RC-LH core comples from Halorhodospira halochloris

EMDB-36760: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

EMDB-36761: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

PDB-8k0c: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

PDB-8k0d: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

EMDB-37342: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

PDB-8w8d: 
Structural mechanism of inhibition of the Rho transcription termination factor by Rof

EMDB-43931: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

EMDB-43932: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex

PDB-9axa: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

PDB-9axc: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex

EMDB-37104: 
96-nm axonemal repeat with RS1/2/3

EMDB-37111: 
48-nm repeat DMT

EMDB-37114: 
Radial Spoke 1 (RS1)

EMDB-37116: 
RS1 refined with head mask

EMDB-37117: 
Radial Spoke 2 (RS2)

EMDB-37118: 
Radial Spoke 2 (RS2) head

EMDB-37119: 
Radial Spoke 3

EMDB-37120: 
Radial Spoke 3 head

EMDB-37626: 
Cryo-EM structure of SARS-CoV-2 prototype spike protein in complex with hippopotamus ACE2

EMDB-37629: 
Cryo-EM structure of SARS-CoV-2 prototype spike protein receptor-binding domain in complex with hippopotamus ACE2

PDB-8wlo: 
Cryo-EM structure of SARS-CoV-2 prototype spike protein in complex with hippopotamus ACE2

PDB-8wlr: 
Cryo-EM structure of SARS-CoV-2 prototype spike protein receptor-binding domain in complex with hippopotamus ACE2

EMDB-37695: 
Cryo-EM structure of human SIDT1 protein with C1 symmetry at neutral pH

EMDB-37696: 
Cryo-EM structure of human SIDT1 protein with C2 symmetry at neutral pH

EMDB-37697: 
Cryo-EM structure of human SIDT1 protein with C1 symmetry at low pH

EMDB-37698: 
Cryo-EM structure of human SIDT1 protein with C2 symmetry at low pH

PDB-8woq: 
Cryo-EM structure of human SIDT1 protein with C1 symmetry at neutral pH

PDB-8wor: 
Cryo-EM structure of human SIDT1 protein with C2 symmetry at neutral pH

PDB-8wos: 
Cryo-EM structure of human SIDT1 protein with C1 symmetry at low pH

PDB-8wot: 
Cryo-EM structure of human SIDT1 protein with C2 symmetry at low pH
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
