-検索条件
-検索結果
検索 (著者・登録者: espinosa & s)の結果55件中、1から50件目までを表示しています

EMDB-17576: 
SARS-CoV-2 S-protein:D614G mutant in 1-up conformation

EMDB-17578: 
SARS-CoV-2 S protein S:D614G mutant in 3-down with binding site of an entry inhibitor

PDB-8p99: 
SARS-CoV-2 S-protein:D614G mutant in 1-up conformation

PDB-8p9y: 
SARS-CoV-2 S protein S:D614G mutant in 3-down with binding site of an entry inhibitor

EMDB-26363: 
Head region of insulin receptor ectodomain (A-isoform) bound to the non-insulin agonist IM459

EMDB-26364: 
Head region of insulin receptor ectodomain (A-isoform) bound to the non-insulin agonist IM462

PDB-7u6d: 
Head region of insulin receptor ectodomain (A-isoform) bound to the non-insulin agonist IM459

PDB-7u6e: 
Head region of insulin receptor ectodomain (A-isoform) bound to the non-insulin agonist IM462

EMDB-26459: 
cryo-EM structure of the rigor state wild type myosin-15-F-actin complex (symmetry expansion and re-centering)
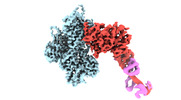
EMDB-26460: 
cryo-EM structure of the ADP state wild type myosin-15-F-actin complex (symmetry expansion and re-centering)
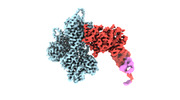
EMDB-26461: 
cryo-EM structure of the ADP state wild type myosin-15-F-actin complex (symmetry expansion)
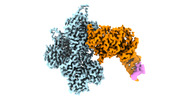
EMDB-26462: 
cryo-EM structure of the rigor state wild type myosin-15-F-actin complex (symmetry expansion)
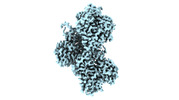
EMDB-26463: 
cryo-EM structure of the ADP state actin filament (symmetry expansion)

EMDB-26464: 
Cryo-EM structure of the rigor state Jordan myosin-15-F-actin complex (symmetry expansion)

PDB-7udt: 
cryo-EM structure of the rigor state wild type myosin-15-F-actin complex (symmetry expansion and re-centering)

PDB-7udu: 
cryo-EM structure of the ADP state wild type myosin-15-F-actin complex (symmetry expansion and re-centering)

EMDB-24321: 
Cryo-EM structure of the ADP state actin filament

EMDB-24322: 
cryo-EM structure of the rigor state wild type myosin-15-F-actin complex

EMDB-24399: 
cryo-EM structure of the ADP state wild type myosin-15-F-actin complex

EMDB-24400: 
Cryo-EM structure of the rigor state Jordan myosin-15-F-actin complex

PDB-7r8v: 
Cryo-EM structure of the ADP state actin filament

PDB-7r91: 
cryo-EM structure of the rigor state wild type myosin-15-F-actin complex

PDB-7rb8: 
cryo-EM structure of the ADP state wild type myosin-15-F-actin complex

PDB-7rb9: 
Cryo-EM structure of the rigor state Jordan myosin-15-F-actin complex

EMDB-20843: 
Alpha-E-catenin ABD-F-actin complex

EMDB-20844: 
Metavinculin ABD-F-actin complex

PDB-6upv: 
Alpha-E-catenin ABD-F-actin complex

PDB-6upw: 
Metavinculin ABD-F-actin complex

EMDB-11115: 
Human Picobirnavirus CP VLP

EMDB-11116: 
Human Picobirnavirus Ht-CP VLP

PDB-6z8d: 
Human Picobirnavirus CP VLP

PDB-6z8e: 
Human Picobirnavirus Ht-CP VLP

EMDB-11117: 
Human Picobirnavirus D45-CP VLP

PDB-6z8f: 
Human Picobirnavirus D45-CP VLP

EMDB-10284: 
Pseudomonas aeruginosa 70s ribosome from an aminoglycoside resistant clinical isolate

PDB-6spf: 
Pseudomonas aeruginosa 70s ribosome from an aminoglycoside resistant clinical isolate

EMDB-10280: 
Pseudomonas aeruginosa 50s ribosome from a clinical isolate with a mutation in uL6

EMDB-10281: 
Pseudomonas aeruginosa 30s ribosome from an aminoglycoside resistant clinical isolate

EMDB-10282: 
Pseudomonas aeruginosa 50s ribosome from a clinical isolate

EMDB-10283: 
Pseudomonas aeruginosa 30s ribosome from a clinical isolate

EMDB-10285: 
Pseudomonas aeruginosa 70s ribosome from a clinical isolate

PDB-6spb: 
Pseudomonas aeruginosa 50s ribosome from a clinical isolate with a mutation in uL6

PDB-6spc: 
Pseudomonas aeruginosa 30s ribosome from an aminoglycoside resistant clinical isolate

PDB-6spd: 
Pseudomonas aeruginosa 50s ribosome from a clinical isolate

PDB-6spe: 
Pseudomonas aeruginosa 30s ribosome from a clinical isolate

PDB-6spg: 
Pseudomonas aeruginosa 70s ribosome from a clinical isolate

EMDB-0360: 
Saccharomyces cerevisiae spliceosomal E complex (UBC4)

EMDB-0361: 
Saccharomyces cerevisiae spliceosomal E complex (ACT1)

PDB-6n7p: 
S. cerevisiae spliceosomal E complex (UBC4)

PDB-6n7r: 
Saccharomyces cerevisiae spliceosomal E complex (ACT1)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
