-検索条件
-検索結果
検索 (著者・登録者: choi & m)の結果248件中、1から50件目までを表示しています

EMDBエントリ 画像なし
EMDB-41849: 
Structure of 310-18A5 Fab in complex with A/Solomon Islands/3/2006(H1N1) influenza virus hemagglutinin

EMDB-37593: 
Vibrio vulnificus MARTX effector duet (RDTND-RID) complexed with human Rac1 Q61L and calmodulin

EMDB-39534: 
Cryo-EM structure of the human ABCB6 in complex with Cd(II):GSH
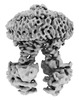
EMDB-39535: 
Cryo-EM structure of the human ABCB6 in complex with Cd(II):Phytochelatin 2

PDB-8yr3: 
Cryo-EM structure of the human ABCB6 in complex with Cd(II):GSH

PDB-8yr4: 
Cryo-EM structure of the human ABCB6 in complex with Cd(II):Phytochelatin 2

EMDB-29659: 
CryoEM structure of nuclear GAPDH under 8h Oxidative Stress

EMDB-29660: 
CryoEM structure of cytosolic GAPDH under 8h Oxidative Stress

EMDB-29661: 
CryoEM structure of cytosolic GAPDH under 8h Oxidative Stress, class2

EMDB-29662: 
CryoEM structure of nuclear GAPDH under 24h Oxidative Stress

EMDB-29663: 
CryoEM structure of cytoplasmic GAPDH under 24h Oxidative Stress

EMDB-29664: 
CryoEM structure of wild-type GAPDH

EMDB-35010: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

EMDB-35770: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Consensus map)

EMDB-35772: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Receptor-focused map)

EMDB-35773: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (G protein-focused map)
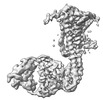
EMDB-35971: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL
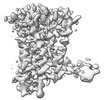
EMDB-37336: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

PDB-8hti: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

PDB-8j46: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL

PDB-8w77: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

EMDB-40699: 
SARS-CoV-2 replication-transcription complex bound to nsp9 and UMPCPP, as a pre-catalytic NMPylation intermediate

EMDB-40707: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9, as a noncatalytic RNA-nsp9 binding mode

EMDB-40708: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9 and GDP-betaS, as a pre-catalytic deRNAylation/mRNA capping intermediate

PDB-8sq9: 
SARS-CoV-2 replication-transcription complex bound to nsp9 and UMPCPP, as a pre-catalytic NMPylation intermediate

PDB-8sqj: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9, as a noncatalytic RNA-nsp9 binding mode

PDB-8sqk: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9 and GDP-betaS, as a pre-catalytic deRNAylation/mRNA capping intermediate

EMDB-28823: 
Phi-29 partially-expanded fiberless prohead

PDB-8f2n: 
Phi-29 partially-expanded fiberless prohead

EMDB-28820: 
phi-29 prohead MCP gp8 penton maturation intermediate, with associated scaffold gp7 tetramer

EMDB-28821: 
bacteriophage phi-29 MCP gp-8 penton in intermediate maturation state, with associated scaffold gp7 dimer

EMDB-28822: 
Phi-29 scaffolding protein bound to intermediate-state MCP

EMDB-28824: 
Phi-29 expanded, DNA-packaged fiberless prohead

EMDB-29013: 
96nm doublet microtubule repeat from wild type mouse sperm

EMDB-28606: 
96nm doublet microtubule repeat from LRRC23-KO mouse sperm

EMDB-36937: 
post-occluded structure of human ABCB6 W546A mutant (ADP/VO4-bound)

EMDB-36938: 
Outward-facing structure of human ABCB6 W546A mutant (ADP/VO4-bound)

PDB-8k7b: 
post-occluded structure of human ABCB6 W546A mutant (ADP/VO4-bound)

PDB-8k7c: 
Outward-facing structure of human ABCB6 W546A mutant (ADP/VO4-bound)
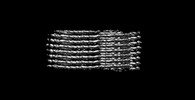
EMDB-40305: 
Cryo-EM structure of insulin amyloid-like fibril that is composed of two antiparallel protofilaments

PDB-8sbd: 
Cryo-EM structure of insulin amyloid-like fibril that is composed of two antiparallel protofilaments

EMDB-33734: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

PDB-7yc5: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

EMDB-34648: 
CryoEM structure of Helicobacter pylori UreFD/urease complex

EMDB-34659: 
CryoEM Structure of Klebsiella pneumoniae UreD/urease complex

PDB-8hc1: 
CryoEM structure of Helicobacter pylori UreFD/urease complex

PDB-8hcn: 
CryoEM Structure of Klebsiella pneumoniae UreD/urease complex

EMDB-34350: 
Legionella pneumophila Deubiquitinase, LotA(7-544) apo state
EMDB-33984: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi

EMDB-33985: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
