-検索条件
-検索結果
検索 (著者・登録者: chiu & w)の結果809件中、1から50件目までを表示しています

EMDB-46646: 
HIV-1 BaL Env in complex with CD4 mimetic CJF-III-288 and 17b IgG

EMDB-41038: 
2.07 Angstrom CryoEM Structure of Heavy Chain Apoferritin from Mus Musculus From 200kV Microscope

PDB-8t4q: 
2.07 Angstrom CryoEM Structure of Heavy Chain Apoferritin from Mus Musculus From 200kV Microscope

EMDB-42603: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/hexamer)

EMDB-42625: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC804515)

EMDB-42626: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/up)

EMDB-42627: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/down)

EMDB-44748: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/dodecamer)

PDB-8uv2: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/hexamer)

PDB-8uvo: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC804515)

PDB-8uvp: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/up)

PDB-8uvq: 
Human p97/VCP R155H mutant structure with a triazole inhibitor (NSC819701/down)

PDB-9boq: 
Human p97/VCP structure with a triazole inhibitor (NSC799462/dodecamer)

EMDB-43385: 
Cryo-EM map of close dodecameric CaMKII beta holoenzyme T287A T306A T307A

EMDB-40873: 
Cryo-EM structure of tetradecameric hub domain of CaMKII alpha

EMDB-41070: 
Cryo-EM structure of tetradecameric CaMKII beta holoenzyme T287A T306A T307A

EMDB-41077: 
Cryo-EM structure of dodecameric CaMKII beta holoenzyme T287A T306A T307A

PDB-8syg: 
Cryo-EM structure of tetradecameric hub domain of CaMKII alpha

PDB-8t6k: 
Cryo-EM structure of tetradecameric CaMKII beta holoenzyme T287A T306A T307A

PDB-8t6q: 
Cryo-EM structure of dodecameric CaMKII beta holoenzyme T287A T306A T307A

EMDB-40955: 
Cryo-EM structure of dodecameric hub domain of CaMKII alpha

EMDB-40956: 
Cryo-EM structure of tetradecameric hub domain of CaMKII beta

EMDB-40957: 
Cryo-EM structure of dodecameric hub domain of CaMKII beta

PDB-8t15: 
Cryo-EM structure of dodecameric hub domain of CaMKII alpha

PDB-8t17: 
Cryo-EM structure of tetradecameric hub domain of CaMKII beta

PDB-8t18: 
Cryo-EM structure of dodecameric hub domain of CaMKII beta

EMDB-36760: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

EMDB-36761: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

PDB-8k0c: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

PDB-8k0d: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

EMDB-36849: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

PDB-8k3c: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

PDB-8sjy: 
Structure of lens aquaporin-0 array in sphingomyelin/cholesterol bilayer (1SM:2Chol)

PDB-8sjx: 
Structure of lens aquaporin-0 array in sphingomyelin/cholesterol bilayer (2SM:1Chol)

EMDB-41637: 
Asymmetric cryoEM reconstruction of Mayaro virus

EMDB-28979: 
Cryo-EM structure of Chikungunya virus asymmetric unit

EMDB-41096: 
Cryo-electron tomography of Chikungunya virus pentamer structure

EMDB-41631: 
Cryo-EM structure of Chikungunya virus with asymmetric reconstruction

PDB-8fcg: 
Cryo-EM structure of Chikungunya virus asymmetric unit

EMDB-42820: 
SARS-CoV-2 5' proximal stem-loop 5 and 6 with SL5b extended

EMDB-28138: 
3D reconstruction of the apical complex of Plasmodium falciparum (3D7) free merozoite

EMDB-28141: 
3D reconstruction of the apical complex of Plasmodium falciparum (3D7) free merozoite
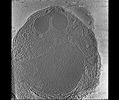
EMDB-28142: 
3D Reconstruction of Plasmodium falciparum (3D7) free merozoite

EMDB-36062: 
Asfv topoisomerase 2 - apo conformer Ia

EMDB-36063: 
Asfv topoisomerase 2 - apo conformer Ib

EMDB-36064: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIa

EMDB-36065: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIb

EMDB-36066: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIa

EMDB-36067: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIb

EMDB-36116: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide (EDI-1)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
