[English] 日本語
 Yorodumi
Yorodumi- EMDB-36849: Nipah virus Attachment glycoprotein with 41-6 antibody fragment -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Nipah virus Attachment glycoprotein with 41-6 antibody fragment | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Nipah virus / Attachment glycoprotein / tetramer complex / Neutralizing antibody / VIRAL PROTEIN / VIRAL PROTEIN-IMMUNE SYSTEM complex | |||||||||
| Function / homology |  Function and homology information Function and homology informationmembrane fusion involved in viral entry into host cell / exo-alpha-sialidase activity / clathrin-dependent endocytosis of virus by host cell / host cell surface receptor binding / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / identical protein binding / membrane Similarity search - Function | |||||||||
| Biological species |  Henipavirus nipahense / Henipavirus nipahense /  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.88 Å | |||||||||
 Authors Authors | Sun MM | |||||||||
| Funding support |  China, 1 items China, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2024 Journal: Nat Commun / Year: 2024Title: Potent human neutralizing antibodies against Nipah virus derived from two ancestral antibody heavy chains. Authors: Li Chen / Mengmeng Sun / Huajun Zhang / Xinghai Zhang / Yanfeng Yao / Ming Li / Kangyin Li / Pengfei Fan / Haiwei Zhang / Ye Qin / Zhe Zhang / Entao Li / Zhen Chen / Wuxiang Guan / Shanshan ...Authors: Li Chen / Mengmeng Sun / Huajun Zhang / Xinghai Zhang / Yanfeng Yao / Ming Li / Kangyin Li / Pengfei Fan / Haiwei Zhang / Ye Qin / Zhe Zhang / Entao Li / Zhen Chen / Wuxiang Guan / Shanshan Li / Changming Yu / Kaiming Zhang / Rui Gong / Sandra Chiu /  Abstract: Nipah virus (NiV) is a World Health Organization priority pathogen and there are currently no approved drugs for clinical immunotherapy. Through the use of a naïve human phage-displayed Fab library, ...Nipah virus (NiV) is a World Health Organization priority pathogen and there are currently no approved drugs for clinical immunotherapy. Through the use of a naïve human phage-displayed Fab library, two neutralizing antibodies (NiV41 and NiV42) targeting the NiV receptor binding protein (RBP) were identified. Following affinity maturation, antibodies derived from NiV41 display cross-reactivity against both NiV and Hendra virus (HeV), whereas the antibody based on NiV42 is only specific to NiV. Results of immunogenetic analysis reveal a correlation between the maturation of antibodies and their antiviral activity. In vivo testing of NiV41 and its mature form (41-6) show protective efficacy against a lethal NiV challenge in hamsters. Furthermore, a 2.88 Å Cryo-EM structure of the tetrameric RBP and antibody complex demonstrates that 41-6 blocks the receptor binding interface. These findings can be beneficial for the development of antiviral drugs and the design of vaccines with broad spectrum against henipaviruses. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_36849.map.gz emd_36849.map.gz | 483.4 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-36849-v30.xml emd-36849-v30.xml emd-36849.xml emd-36849.xml | 17.6 KB 17.6 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_36849.png emd_36849.png | 65.3 KB | ||
| Filedesc metadata |  emd-36849.cif.gz emd-36849.cif.gz | 6.2 KB | ||
| Others |  emd_36849_half_map_1.map.gz emd_36849_half_map_1.map.gz emd_36849_half_map_2.map.gz emd_36849_half_map_2.map.gz | 474.6 MB 474.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36849 http://ftp.pdbj.org/pub/emdb/structures/EMD-36849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36849 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36849 | HTTPS FTP |
-Related structure data
| Related structure data |  8k3cMC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_36849.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_36849.map.gz / Format: CCP4 / Size: 512 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.82 Å | ||||||||||||||||||||||||||||||||||||
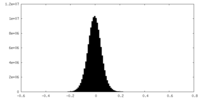
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #2
| File | emd_36849_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
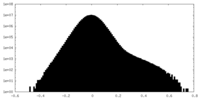
| Density Histograms |
-Half map: #1
| File | emd_36849_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
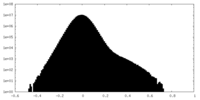
| Density Histograms |
- Sample components
Sample components
-Entire : Cryo-EM structure of complex of attachment glycoprotein G with 41...
| Entire | Name: Cryo-EM structure of complex of attachment glycoprotein G with 41-6 Fab fragment |
|---|---|
| Components |
|
-Supramolecule #1: Cryo-EM structure of complex of attachment glycoprotein G with 41...
| Supramolecule | Name: Cryo-EM structure of complex of attachment glycoprotein G with 41-6 Fab fragment type: complex / ID: 1 / Parent: 0 / Macromolecule list: all |
|---|---|
| Source (natural) | Organism:  Henipavirus nipahense Henipavirus nipahense |
-Macromolecule #1: Light chain of 41-6 Fab fragment
| Macromolecule | Name: Light chain of 41-6 Fab fragment / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 22.844219 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: QSVVTQPPSV SAAPGQKVTI SCSGSSSNIG TNHLSWYQQV PGTAPKLLIY DNNKRPSGIP DRFSGSKSGT SASLAISGLQ SEDEADYYC ATWDDSLHAW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP ...String: QSVVTQPPSV SAAPGQKVTI SCSGSSSNIG TNHLSWYQQV PGTAPKLLIY DNNKRPSGIP DRFSGSKSGT SASLAISGLQ SEDEADYYC ATWDDSLHAW VFGGGTKLTV LGQPKAAPSV TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV K AGVETTTP SKQSNNKYAA SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS |
-Macromolecule #2: Heavy chain of 41-6 Fab fragments
| Macromolecule | Name: Heavy chain of 41-6 Fab fragments / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 25.312264 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: EVQLVQSGGG LVQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSA ISGSGGSTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAE DTAVYYCARD REDIVVVPAP RGYYYYYYMD VWGQGTTVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL V KDYFPEPV ...String: EVQLVQSGGG LVQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSA ISGSGGSTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAE DTAVYYCARD REDIVVVPAP RGYYYYYYMD VWGQGTTVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL V KDYFPEPV TVSWNSGALT SGVHTFPAVL QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKK VEPKSCDKTS |
-Macromolecule #3: Glycoprotein G
| Macromolecule | Name: Glycoprotein G / type: protein_or_peptide / ID: 3 / Number of copies: 2 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Henipavirus nipahense Henipavirus nipahense |
| Molecular weight | Theoretical: 52.941188 KDa |
| Recombinant expression | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Sequence | String: ISQSTASINE NVNEKCKFTL PPLKIHECNI SCPNPLPFRE YRPQTEGVSN LVGLPNNICL QKTSNQILKP KLISYTLPVV GQSGTCITD PLLAMDEGYF AYSHLERIGS CSRGVSKQRI IGVGEVLDRG DEVPSLFMTN VWTPPNPNTV YHCSAVYNNE F YYVLCAVS ...String: ISQSTASINE NVNEKCKFTL PPLKIHECNI SCPNPLPFRE YRPQTEGVSN LVGLPNNICL QKTSNQILKP KLISYTLPVV GQSGTCITD PLLAMDEGYF AYSHLERIGS CSRGVSKQRI IGVGEVLDRG DEVPSLFMTN VWTPPNPNTV YHCSAVYNNE F YYVLCAVS TVGDPILNST YWSGSLMMTR LAVKPKSNGG GYNQHQLALR SIEKGRYDKV MPYGPSGIKQ GDTLYFPAVG FL VRTEFKY NDSNCPITKC QYSKPENCRL SMGIRPNSHY ILRSGLLKYN LSDGENPKVV FIEISDQRLS IGSPSKIYDS LGQ PVFYQA SFSWDTMIKF GDVLTVNPLV VNWRNNTVIS RPGQSQCPRF NTCPEICWEG VYNDAFLIDR INWISAGVFL DSNQ TAENP VFTVFKDNEI LYRAQLASED TNAQKTITNC FLLKNKIWCI SLVEIYDTGD NVIRPKLFAV KIPEQC UniProtKB: Glycoprotein G |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.16 mg/mL |
|---|---|
| Buffer | pH: 8 |
| Grid | Model: Quantifoil R2/1 / Material: COPPER / Mesh: 200 |
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Digitization - Dimensions - Width: 4092 pixel / Digitization - Dimensions - Height: 5760 pixel / Number real images: 9103 / Average exposure time: 3.0 sec. / Average electron dose: 51.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 3.2 µm / Nominal defocus min: 2.0 µm / Nominal magnification: 105000 |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)