-検索条件
-検索結果
検索 (著者・登録者: artur & b)の結果93件中、1から50件目までを表示しています

EMDB-18313: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

EMDB-18314: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

EMDB-18315: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-18317: 
Retron-Eco1 filament (2 segments)

EMDB-19792: 
Retron-Eco1 -1 turn mutant filament with ADP-ribosylated Effector (Consensus refinement)

EMDB-19793: 
Retron-Eco1 filament with ADP-ribosylated Effector (Consensus refinement)

PDB-8qbk: 
Retron-Eco1 filament with ADP-ribosylated Effector (local map with 1 segment)

PDB-8qbl: 
Retron-Eco1 filament with inactive effector (E106A, 2 segments)

PDB-8qbm: 
Retron-Eco1 filament with ADP-ribosylated Effector (full map with 2 segments)

EMDB-16466: 
The structural architecture of alpha-synuclein oligomer

EMDB-16528: 
3D reconstruction of alpha-synuclein oligomer-PSMa3 complex

EMDB-40032: 
Structure of Trypanosoma (MDH)4-(Pex5)2, distal conformation

PDB-8gh3: 
Structure of Trypanosoma (MDH)4-(Pex5)2, distal conformation
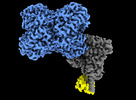
EMDB-40056: 
Structure of Trypanosoma docking complex

PDB-8gi0: 
Structure of Trypanosoma docking complex

EMDB-40003: 
Structure of Trypanosoma (MDH)4-Pex5, close conformation

PDB-8ggd: 
Structure of Trypanosoma (MDH)4-Pex5, close conformation
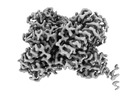
EMDB-40053: 
Consensus cryo-EM reconstruction of Trypanosoma Docking complex

EMDB-40008: 
Structure of Trypanosoma (MDH)4-PEX5, distal conformation

PDB-8ggh: 
Structure of Trypanosoma (MDH)4-PEX5, distal conformation
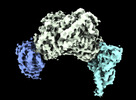
EMDB-40031: 
Structure of Trypanosoma (MDH)4-(Pex5)2, close conformation

PDB-8gh2: 
Structure of Trypanosoma (MDH)4-(Pex5)2, close conformation

EMDB-40055: 
Focused map of Pex region of Trypanosoma docking complex, (MDH)4-(Pex5)1-(Pex14)1

EMDB-18520: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

EMDB-18521: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

EMDB-18522: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

EMDB-18523: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

PDB-8qo2: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

PDB-8qo3: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

PDB-8qo4: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

PDB-8qo5: 
Conserved Structures and Dynamics in 5-Proximal Regions of Betacoronavirus RNA Genomes

EMDB-17195: 
CryoEM reconstruction of a TRAP protein cage made out of 12 11-membered rings

EMDB-17196: 
Structure of the protein cage composed of 12 identical 12-membered protein rings

EMDB-28022: 
Adeno-associated virus type 2 VLP displaying M2 peptide
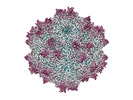
EMDB-28040: 
Adeno-associated virus type 2 VLP displaying Linker peptide

EMDB-14727: 
3D reconstruction of the cylindrical assembly of DnaJA2 by imposing D5 symmetry

EMDB-14729: 
3D reconstruction of the cylindrical assembly of DnaJA2 without symmetry imposition

EMDB-14736: 
3D reconstruction of the cylindrical assembly of DnaJA2 delta G/F by imposing D5 symmetry

PDB-7zhs: 
3D reconstruction of the cylindrical assembly of DnaJA2 delta G/F by imposing D5 symmetry

EMDB-14706: 
3D reconstruction of the cylindrical assembly of DnaJA2 delta G/F without symmetry imposition
EMDB-16016: 
ISDra2 TnpB in complex with reRNA

PDB-8bf8: 
ISDra2 TnpB in complex with reRNA

EMDB-28655: 
ISDra2 TnpB in complex with reRNA and cognate DNA, conformation 2 (RuvC domain unresolved)
EMDB-28656: 
ISDra2 TnpB in complex with reRNA and cognate DNA, conformation 1 (RuvC domain resolved)

PDB-8ex9: 
ISDra2 TnpB in complex with reRNA and cognate DNA, conformation 2 (RuvC domain unresolved)

PDB-8exa: 
ISDra2 TnpB in complex with reRNA and cognate DNA, conformation 1 (RuvC domain resolved)

EMDB-15052: 
CryoEM structure of DHS-eIF5A1 complex

PDB-8a0e: 
CryoEM structure of DHS-eIF5A1 complex

EMDB-26005: 
Structure of the Inmazeb cocktail and resistance to escape against Ebola virus

PDB-7tn9: 
Structure of the Inmazeb cocktail and resistance to escape against Ebola virus
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
