+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | CryoEM structure of DHS-eIF5A1 complex | |||||||||
 Map data Map data | main map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Hypusination / transferase / posttranslational modification | |||||||||
| Function / homology |  Function and homology information Function and homology informationpositive regulation of translational termination / peptidyl-hypusine biosynthetic process / deoxyhypusine synthase / Hypusine synthesis from eIF5A-lysine / deoxyhypusine synthase activity / spermidine metabolic process / spermidine catabolic process / positive regulation of translational elongation / translation elongation factor activity / translation initiation factor activity ...positive regulation of translational termination / peptidyl-hypusine biosynthetic process / deoxyhypusine synthase / Hypusine synthesis from eIF5A-lysine / deoxyhypusine synthase activity / spermidine metabolic process / spermidine catabolic process / positive regulation of translational elongation / translation elongation factor activity / translation initiation factor activity / positive regulation of T cell proliferation / glucose homeostasis / ribosome binding / translation / positive regulation of cell population proliferation / endoplasmic reticulum membrane / RNA binding / identical protein binding / cytoplasm / cytosol Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.8 Å | |||||||||
 Authors Authors | Wator E / Wilk P / Biela AP / Rawski M / Grudnik P | |||||||||
| Funding support |  Poland, 2 items Poland, 2 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2023 Journal: Nat Commun / Year: 2023Title: Cryo-EM structure of human eIF5A-DHS complex reveals the molecular basis of hypusination-associated neurodegenerative disorders. Authors: Elżbieta Wątor / Piotr Wilk / Artur Biela / Michał Rawski / Krzysztof M Zak / Wieland Steinchen / Gert Bange / Sebastian Glatt / Przemysław Grudnik /   Abstract: Hypusination is a unique post-translational modification of the eukaryotic translation factor 5A (eIF5A) that is essential for overcoming ribosome stalling at polyproline sequence stretches. The ...Hypusination is a unique post-translational modification of the eukaryotic translation factor 5A (eIF5A) that is essential for overcoming ribosome stalling at polyproline sequence stretches. The initial step of hypusination, the formation of deoxyhypusine, is catalyzed by deoxyhypusine synthase (DHS), however, the molecular details of the DHS-mediated reaction remained elusive. Recently, patient-derived variants of DHS and eIF5A have been linked to rare neurodevelopmental disorders. Here, we present the cryo-EM structure of the human eIF5A-DHS complex at 2.8 Å resolution and a crystal structure of DHS trapped in the key reaction transition state. Furthermore, we show that disease-associated DHS variants influence the complex formation and hypusination efficiency. Hence, our work dissects the molecular details of the deoxyhypusine synthesis reaction and reveals how clinically-relevant mutations affect this crucial cellular process. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_15052.map.gz emd_15052.map.gz | 59.7 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-15052-v30.xml emd-15052-v30.xml emd-15052.xml emd-15052.xml | 20.7 KB 20.7 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_15052_fsc.xml emd_15052_fsc.xml | 10.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_15052.png emd_15052.png | 52.8 KB | ||
| Filedesc metadata |  emd-15052.cif.gz emd-15052.cif.gz | 6.5 KB | ||
| Others |  emd_15052_half_map_1.map.gz emd_15052_half_map_1.map.gz emd_15052_half_map_2.map.gz emd_15052_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-15052 http://ftp.pdbj.org/pub/emdb/structures/EMD-15052 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15052 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-15052 | HTTPS FTP |
-Related structure data
| Related structure data |  8a0eMC  7a6sC  7a6tC  8a0fC  8a0gC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_15052.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_15052.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | main map | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: half map A
| File | emd_15052_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half map A | ||||||||||||
| Projections & Slices |
| ||||||||||||
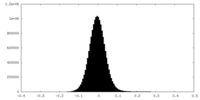
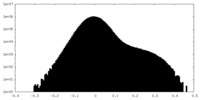
| Density Histograms |
-Half map: half map B
| File | emd_15052_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | half map B | ||||||||||||
| Projections & Slices |
| ||||||||||||
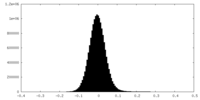
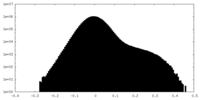
| Density Histograms |
- Sample components
Sample components
-Entire : Complex of human DHS-eIF5A
| Entire | Name: Complex of human DHS-eIF5A |
|---|---|
| Components |
|
-Supramolecule #1: Complex of human DHS-eIF5A
| Supramolecule | Name: Complex of human DHS-eIF5A / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 / Details: monomer of eIF5A bound to homotetramer DHS. |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 181.687 KDa |
-Macromolecule #1: Deoxyhypusine synthase
| Macromolecule | Name: Deoxyhypusine synthase / type: protein_or_peptide / ID: 1 / Number of copies: 2 / Enantiomer: LEVO / EC number: deoxyhypusine synthase |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 41.098461 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GSMEGSLERE APAGALAAVL KHSSTLPPES TQVRGYDFNR GVNYRALLEA FGTTGFQATN FGRAVQQVNA MIEKKLEPLS QDEDQHADL TQSRRPLTSC TIFLGYTSNL ISSGIRETIR YLVQHNMVDV LVTTAGGVEE DLIKCLAPTY LGEFSLRGKE L RENGINRI ...String: GSMEGSLERE APAGALAAVL KHSSTLPPES TQVRGYDFNR GVNYRALLEA FGTTGFQATN FGRAVQQVNA MIEKKLEPLS QDEDQHADL TQSRRPLTSC TIFLGYTSNL ISSGIRETIR YLVQHNMVDV LVTTAGGVEE DLIKCLAPTY LGEFSLRGKE L RENGINRI GNLLVPNENY CKFEDWLMPI LDQMVMEQNT EGVKWTPSKM IARLGKEINN PESVYYWAQK NHIPVFSPAL TD GSLGDMI FFHSYKNPGL VLDIVEDLRL INTQAIFAKC TGMIILGGGV VKHHIANANL MRNGADYAVY INTAQEFDGS DSG ARPDEA VSWGAIRVDA QPVKVYADAS LVFPLLVAET FAQKMDAFMH EKNED UniProtKB: Deoxyhypusine synthase |
-Macromolecule #2: Deoxyhypusine synthase
| Macromolecule | Name: Deoxyhypusine synthase / type: protein_or_peptide / ID: 2 / Number of copies: 2 / Enantiomer: LEVO / EC number: deoxyhypusine synthase |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 41.130527 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GSMEGSLERE APAGALAAVL KHSSTLPPES TQVRGYDFNR GVNYRALLEA FGTTGFQATN FGRAVQQVNA MIEKKLEPLS QDEDQHADL TQSRRPLTSC TIFLGYTSNL ISSGIRETIR YLVQHNMVDV LVTTAGGVEE DLIKCLAPTY LGEFSLRGKE L RENGINRI ...String: GSMEGSLERE APAGALAAVL KHSSTLPPES TQVRGYDFNR GVNYRALLEA FGTTGFQATN FGRAVQQVNA MIEKKLEPLS QDEDQHADL TQSRRPLTSC TIFLGYTSNL ISSGIRETIR YLVQHNMVDV LVTTAGGVEE DLIKCLAPTY LGEFSLRGKE L RENGINRI GNLLVPNENY (CSS)KFEDWLMPI LDQMVMEQNT EGVKWTPSKM IARLGKEINN PESVYYWAQK NHIPVFSP A LTDGSLGDMI FFHSYKNPGL VLDIVEDLRL INTQAIFAKC TGMIILGGGV VKHHIANANL MRNGADYAVY INTAQEFDG SDSGARPDEA VSWGAIRVDA QPVKVYADAS LVFPLLVAET FAQKMDAFMH EKNED UniProtKB: Deoxyhypusine synthase |
-Macromolecule #3: Eukaryotic translation initiation factor 5A
| Macromolecule | Name: Eukaryotic translation initiation factor 5A / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 16.998395 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: GSMADDLDFE TGDAGASATF PMQCSALRKN GFVVLKGRPC KIVEMSTSKT GKHGHAKVHL VGIDIFTGKK YEDICPSTHN MDVPNIKRN DFQLIGIQDG YLSLLQDSGE VREDLRLPEG DLGKEIEQKY DCGEEILITV LSAMTEEAAV AIKAMAK UniProtKB: Eukaryotic translation initiation factor 5A |
-Macromolecule #4: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| Macromolecule | Name: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / type: ligand / ID: 4 / Number of copies: 4 / Formula: NAD |
|---|---|
| Molecular weight | Theoretical: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-Macromolecule #5: SPERMIDINE
| Macromolecule | Name: SPERMIDINE / type: ligand / ID: 5 / Number of copies: 3 / Formula: SPD |
|---|---|
| Molecular weight | Theoretical: 145.246 Da |
| Chemical component information |  ChemComp-SPD: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.12 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 9.3 Component:
| |||||||||
| Grid | Model: Quantifoil R2/2 / Material: COPPER / Mesh: 400 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 70 sec. / Pretreatment - Atmosphere: OTHER | |||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 278 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 40.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 2.7 mm / Nominal defocus max: 3.5 µm / Nominal defocus min: 0.5 µm |
| Sample stage | Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)