-検索条件
-検索結果
検索 (著者・登録者: yamaguchi & t)の結果70件中、1から50件目までを表示しています

EMDB-34791: 
Doublet microtubule of zebrafish sperm axoneme, WT

EMDB-34792: 
Radial spoke 1 of zebrafish sperm axoneme, WT

EMDB-34793: 
Radial spoke 2 of zebrafish sperm axoneme, WT

EMDB-34794: 
Radial spoke 3 of zebrafish sperm axoneme, WT
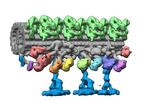
EMDB-34795: 
Doublet microtubule of zebrafish sperm axoneme, calaxin-/-, OAD+ class
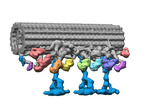
EMDB-34796: 
Doublet microtubule of zebrafish sperm axoneme, calaxin-/-, OAD- class
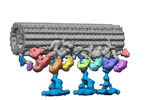
EMDB-34797: 
Doublet microtubule of zebrafish sperm axoneme, armc4-/-

EMDB-34798: 
Outer arm dynein of zebrafish sperm axoneme, WT

EMDB-34799: 
Outer arm dynein of zebrafish sperm axoneme, WT, refined focusing on the docking complex

EMDB-34800: 
Outer arm dynein of zebrafish sperm axoneme, WT, 1 mM calcium condition, refined focusing on the docking complex

EMDB-34801: 
Outer arm dynein of zebrafish sperm axoneme, calaxin-/-, refined focusing on the docking complex

EMDB-34802: 
Outer arm dynein of zebrafish sperm axoneme, calaxin-/-, incubated with recombinant Calaxin protein, refined focusing on the docking complex

EMDB-33200: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-ordered form)
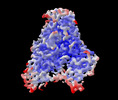
EMDB-33201: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-disordered form)
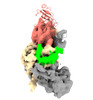
EMDB-33298: 
Cryo-EM map of apo-DNMT1 (aa:351-1616)
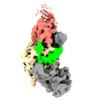
EMDB-33299: 
Cryo-EM map of DNMT1 (aa:351-1616) in complex with ubiquitinated H3

PDB-7xi9: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-ordered form)

PDB-7xib: 
Cryo-EM structure of human DNMT1 (aa:351-1616) in complex with ubiquitinated H3 and hemimethylated DNA analog (CXXC-disordered form)
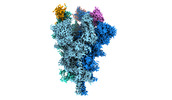
EMDB-32078: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P86
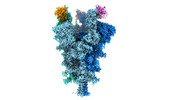
EMDB-32079: 
Cryo-EM structure of the SARS-CoV-2 spike protein (3-up RBD) bound to neutralizing nanobodies P86
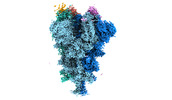
EMDB-32080: 
Cryo-EM structure of the SARS-CoV-2 spike protein (1-up RBD) bound to neutralizing nanobodies P17
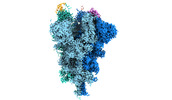
EMDB-32081: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P17

PDB-7vq0: 
Cryo-EM structure of the SARS-CoV-2 spike protein (2-up RBD) bound to neutralizing nanobodies P86
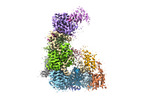
EMDB-31741: 
Structure of the Dicer-2-R2D2 heterodimer
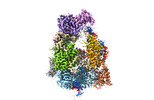
EMDB-31742: 
Structure of the Dicer-2-R2D2 heterodimer bound to small RNA duplex

PDB-7v6b: 
Structure of the Dicer-2-R2D2 heterodimer

PDB-7v6c: 
Structure of the Dicer-2-R2D2 heterodimer bound to small RNA duplex

EMDB-30398: 
CryoEM structure of S.typhimurium flagellar LP ring

EMDB-30409: 
CryoEM structure of S.typhimurium flagellar hook-basal body (HBB) complex

PDB-7clr: 
CryoEM structure of S.typhimurium flagellar LP ring

EMDB-30007: 
Cryo-EM structure of phosphoketolase from Bifidobacterium longum

PDB-6lxv: 
Cryo-EM structure of phosphoketolase from Bifidobacterium longum

EMDB-0730: 
CryoEM map and model of Nitrite Reductase at pH 6.2

EMDB-0731: 
CryoEM map and model of Nitrite Reductase at pH 8.1

PDB-6knf: 
CryoEM map and model of Nitrite Reductase at pH 6.2

PDB-6kng: 
CryoEM map and model of Nitrite Reductase at pH 8.1

EMDB-0980: 
CryoEM structure of S.typhimurium R-type straight flagellar filament made of FljB (A461V)

EMDB-9896: 
CryoEM structure of S.typhimurium R-type flagellar filament made of FljB (A461V) without domain D3 by masking out

PDB-6jy0: 
CryoEM structure of S.typhimurium R-type straight flagellar filament made of FljB (A461V)

EMDB-0809: 
Cryo-electron tomography of Candidatus Prometheoarchaeum syntrophicum strain MK-D1

EMDB-0852: 
Cryo-electron tomography of Candidatus Prometheoarchaeum syntrophicum strain MK-D1

EMDB-9590: 
structure of leucine dehydrogenase from Geobacillus stearothermophilus by cryo-EM

EMDB-9592: 
Structure of NAD+-bound leucine dehydrogenase from Geobacillus stearothermophilus by cryo-EM

PDB-6acf: 
structure of leucine dehydrogenase from Geobacillus stearothermophilus by cryo-EM

PDB-6ach: 
Structure of NAD+-bound leucine dehydrogenase from Geobacillus stearothermophilus by cryo-EM

EMDB-6954: 
Doublet microtubule of zebrafish sperm axoneme, WT

EMDB-6955: 
Doublet microtubule of zebrafish sperm axoneme, pih1d1_null mutant

EMDB-6956: 
Doublet microtubule of zebrafish sperm axoneme, pih1d2_null mutant

EMDB-6957: 
Doublet microtubule of zebrafish sperm axoneme, ktu_null mutant

EMDB-6958: 
Doublet microtubule of zebrafish sperm axoneme, twister_null mutant
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
