+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7b2c | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
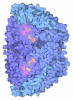
| タイトル | Crystal structure of the ethyl-coenzyme M reductase from Candidatus Ethanoperedens thermophilum gassed with xenon | |||||||||
 要素 要素 | (Ethyl-Coenzyme M reductase ...) x 3 | |||||||||
 キーワード キーワード | TRANSFERASE / Ethyl-CoM reductase / Methyl-CoM reductase / xenon-derivatization / ethane-oxidizers / F430-cofactor / post-translational modification / gas channel / coenzyme M / coenzyme B / true atomic resolution / thermophile / archaea. | |||||||||
| 機能・相同性 | 1-THIOETHANESULFONIC ACID / : / Coenzyme B / Dimethylated-F430 cofactor / Chem-UWT / XENON 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.8 Å 分子置換 / 解像度: 1.8 Å | |||||||||
 データ登録者 データ登録者 | Wagner, T. / Lemaire, O.N. / Engilberge, S. | |||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Crystal structure of a key enzyme for anaerobic ethane activation. 著者: Hahn, C.J. / Lemaire, O.N. / Kahnt, J. / Engilberge, S. / Wegener, G. / Wagner, T. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7b2c.cif.gz 7b2c.cif.gz | 1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7b2c.ent.gz pdb7b2c.ent.gz | 867.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7b2c.json.gz 7b2c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7b2c_validation.pdf.gz 7b2c_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7b2c_full_validation.pdf.gz 7b2c_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  7b2c_validation.xml.gz 7b2c_validation.xml.gz | 114.6 KB | 表示 | |
| CIF形式データ |  7b2c_validation.cif.gz 7b2c_validation.cif.gz | 173.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b2/7b2c https://data.pdbj.org/pub/pdb/validation_reports/b2/7b2c ftp://data.pdbj.org/pub/pdb/validation_reports/b2/7b2c ftp://data.pdbj.org/pub/pdb/validation_reports/b2/7b2c | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Ethyl-Coenzyme M reductase ... , 3種, 6分子 ADBECF
| #1: タンパク質 | 分子量: 66344.344 Da / 分子数: 2 / 変異: wild-type / 由来タイプ: 天然 詳細: Seven post-translational modifications exist in the subunit: N1-methylhistidine291 5(S)-methylarginine305 S-methylcysteine354 3-methylisoleucine377 2(S)-methylglutamine445 Thioglycine490 N2-methylhistidine491 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Culture enrichment / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 49874.453 Da / 分子数: 2 / 変異: wild-type / 由来タイプ: 天然 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Culture enrichment / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase #3: タンパク質 | 分子量: 30578.789 Da / 分子数: 2 / 変異: wild-type / 由来タイプ: 天然 由来: (天然)  Candidatus Ethanoperedens thermophilum (古細菌) Candidatus Ethanoperedens thermophilum (古細菌)細胞株: / / 器官: / / Plasmid details: Culture enrichment / Variant: / / 組織: / / 参照: coenzyme-B sulfoethylthiotransferase |
|---|
-非ポリマー , 12種, 2405分子 



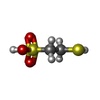



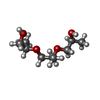














| #4: 化合物 | | #5: 化合物 | ChemComp-GOL / #6: 化合物 | ChemComp-NA / #7: 化合物 | #8: 化合物 | #9: 化合物 | ChemComp-K / #10: 化合物 | ChemComp-XE / #11: 化合物 | ChemComp-CL / #12: 化合物 | #13: 化合物 | ChemComp-MG / | #14: 化合物 | ChemComp-TRS / | #15: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.28 Å3/Da / 溶媒含有率: 46.06 % / 解説: Yellow brick of 200 um long |
|---|---|
| 結晶化 | 温度: 293.15 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: Crystals were obtained by initial screening at 20 degree Celsius using the sitting drop method on a 96-Well MRC 2-Drop Crystallization Plates in polystyrene (SWISSCI). The crystallization ...詳細: Crystals were obtained by initial screening at 20 degree Celsius using the sitting drop method on a 96-Well MRC 2-Drop Crystallization Plates in polystyrene (SWISSCI). The crystallization reservoir contained 90 ul of mother liquor, crystallization drop contained a mixture of 0.6 ul protein at 16.22 mg.ml-1 and 0.6 ul precipitant. The crystal was obtained by initial screening using the JBScreen Pentaerythritol screen from Jena Bioscience in a Coy tent under an N2/H2 atmosphere (95:5 %). The crystallization reservoir contained 45 % (w/v) Pentaerythritol Propoxylate (5/4 PO/OH), 100 mM Tris pH 8.5 and 400 mM potassium chloride. Crystal was harvested using a MiTeGen MicroRT loop. In order to protect the crystal during xenon pressurisation, a part of the plastic capillary, usually used for room temperature diffraction experiments, was placed around it. The loop was then mounted in an Oxford Xcell xenon chamber. Xenon pressure was gradually increased using a manual pump to reach 25 bars. After 7 min, pressure was slowly released and the crystal immediately plunged in liquid nitrogen. PH範囲: 7.5 - 8.5 / Temp details: / |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X06DA / 波長: 1.54981 Å / ビームライン: X06DA / 波長: 1.54981 Å |
| 検出器 | タイプ: DECTRIS PILATUS 2M-F / 検出器: PIXEL / 日付: 2019年9月29日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.54981 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→60.87 Å / Num. obs: 232772 / % possible obs: 98.1 % / 冗長度: 6 % / CC1/2: 0.997 / Rmerge(I) obs: 0.105 / Rpim(I) all: 0.046 / Net I/σ(I): 11.7 |
| 反射 シェル | 解像度: 1.8→1.84 Å / 冗長度: 4.5 % / Rmerge(I) obs: 0.652 / Mean I/σ(I) obs: 2 / Num. unique obs: 11640 / CC1/2: 0.739 / Rpim(I) all: 0.331 / % possible all: 94.6 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 7B1S 解像度: 1.8→35.9 Å / Cor.coef. Fo:Fc: 0.907 / Cor.coef. Fo:Fc free: 0.888 / SU R Cruickshank DPI: 0.198 / 交差検証法: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.135 / SU Rfree Blow DPI: 0.118 / SU Rfree Cruickshank DPI: 0.116 詳細: LAST REFINEMENT CYCLES WERE PERFORMED WITH HYDROGENS. HYDROGENS WERE OMITTED IN THE FINAL DEPOSITED MODEL.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 91.96 Å2 / Biso mean: 17.66 Å2 / Biso min: 3 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.22 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.8→35.9 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.8→1.82 Å / Rfactor Rfree error: 0 / Total num. of bins used: 51
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj