+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3nir | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of small protein crambin at 0.48 A resolution | ||||||
 要素 要素 | Crambin | ||||||
 キーワード キーワード | PLANT PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Crambe hispanica (植物) Crambe hispanica (植物) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / ISOMORPHOUS TO PDB ENTRY 1EJG / 解像度: 0.48 Å シンクロトロン / ISOMORPHOUS TO PDB ENTRY 1EJG / 解像度: 0.48 Å | ||||||
 データ登録者 データ登録者 | Schmidt, A. / Teeter, M. / Weckert, E. / Lamzin, V.S. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.F / 年: 2011 ジャーナル: Acta Crystallogr.,Sect.F / 年: 2011タイトル: Crystal structure of small protein crambin at 0.48 A resolution 著者: Schmidt, A. / Teeter, M. / Weckert, E. / Lamzin, V.S. #1:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2000 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2000タイトル: Accurate protein crystallography at ultra-high resolution: valence electron distribution in crambin. 著者: Jelsch, C. / Teeter, M.M. / Lamzin, V.S. / Pichon-Pesme, V. / Blessing, R.H. / Lecomte, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3nir.cif.gz 3nir.cif.gz | 48.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3nir.ent.gz pdb3nir.ent.gz | 36.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3nir.json.gz 3nir.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ni/3nir https://data.pdbj.org/pub/pdb/validation_reports/ni/3nir ftp://data.pdbj.org/pub/pdb/validation_reports/ni/3nir ftp://data.pdbj.org/pub/pdb/validation_reports/ni/3nir | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | 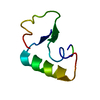 1ejgS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 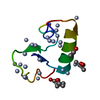
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4738.447 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Crambe hispanica (植物) / 株: SUBSP. ABYSSINICA / 参照: UniProt: P01542 Crambe hispanica (植物) / 株: SUBSP. ABYSSINICA / 参照: UniProt: P01542 | ||||
|---|---|---|---|---|---|
| #2: 化合物 | ChemComp-EOH / #3: 水 | ChemComp-HOH / | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 3 X線回折 / 使用した結晶の数: 3 |
|---|
- 試料調製
試料調製
| 結晶 |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: 60% ethanol in water, pH 7, VAPOR DIFFUSION, SITTING DROP, temperature 293K |
-データ収集
| 回折 |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 |
| ||||||||||||||||||
| 検出器 |
| ||||||||||||||||||
| 放射 |
| ||||||||||||||||||
| 放射波長 |
| ||||||||||||||||||
| 反射 | 解像度: 0.48→20 Å / Num. all: 156860 / Num. obs: 156860 / % possible obs: 97 % / 冗長度: 3.7 % / Biso Wilson estimate: 2.14 Å2 / Rsym value: 0.075 / Net I/σ(I): 9.7 | ||||||||||||||||||
| 反射 シェル | 解像度: 0.48→0.49 Å / Mean I/σ(I) obs: 1.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: ISOMORPHOUS TO PDB ENTRY 1EJG 開始モデル: PDB entry 1EJG 解像度: 0.48→20 Å / Isotropic thermal model: overall / 交差検証法: RFREE INITIALLY, FINAL STAGES NONE / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: Engh & Huber 詳細: INITIAL REFINEMENT WITH REFMAC THEN SHELX, THEN MOPRO FOR MULTIPOLE REFINEMENT. ONLY DURING REFMAC CROSS VALIDATION WITH RFREE. IN THE LAST STAGES OF REFINEMENT THE SUM OF OCCUPANCIES FOR THE ...詳細: INITIAL REFINEMENT WITH REFMAC THEN SHELX, THEN MOPRO FOR MULTIPOLE REFINEMENT. ONLY DURING REFMAC CROSS VALIDATION WITH RFREE. IN THE LAST STAGES OF REFINEMENT THE SUM OF OCCUPANCIES FOR THE MULTIPLE CONFORMERS WAS NOT CONSTRAINED TO 1.00 AND ONLY WEAK STEREOCHEMICAL RESTRAINTS WERE APPLIED. ALTHOUGH OVERALL SUCH REFINEMENT OF THE MULTIPLE CONFORMERS WENT FINE, THERE WAS SOME INSTABILITY - FOR EXAMPLE THE REFINEMENT OF THE TEMPERATURE FACTORS FOR CA AND C ATOMS OF THE THIRD CONFORMER OF TYR A 29 DID NOT CONVERGE AND WERE TRUNCATED TO THE VALUE OF 50. BULK SOLVENT MODELING METHOD USED:BULK SOLVENT ENVELOPE METHOD FC(MOL)+FC(BULK)
| |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 4.12 Å2 | |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 0.48→20 Å
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







