登録情報 データベース : EMDB / ID : EMD-22337タイトル Cryo-EM structure of ATP-bound fully inactive AMPK in complex with Fab and nanobody Structure of AMPK with Fab and nanobody 複合体 : AMPK complex with Fab and nanobody複合体 : AMPKタンパク質・ペプチド : 5'-AMP-activated protein kinase catalytic subunit alpha-1タンパク質・ペプチド : 5'-AMP-activated protein kinase subunit beta-2タンパク質・ペプチド : 5'-AMP-activated protein kinase subunit gamma-1タンパク質・ペプチド : Maltodextrin-binding protein複合体 : Fab, nanobodyタンパク質・ペプチド : Fab light chainタンパク質・ペプチド : Fab heavy chainタンパク質・ペプチド : Nanobodyリガンド : ADENOSINE-5'-TRIPHOSPHATEリガンド : ADENOSINE-5'-DIPHOSPHATEリガンド : ADENOSINE MONOPHOSPHATE機能・相同性 分子機能 ドメイン・相同性 構成要素
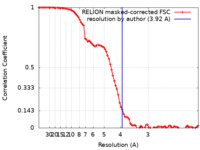
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト) / synthetic construct (人工物) / Escherichia coli K-12 (大腸菌)手法 / / 解像度 : 3.92 Å Yan Y / Murkherjee S / Zhou XE / Xu TH / Xu HE / Kossiakoff AA / Melcher K 資金援助 Organization Grant number 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) R01 GM117372 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) R01 GM129436
ジャーナル : Science / 年 : 2021タイトル : Structure of an AMPK complex in an inactive, ATP-bound state.著者: Yan Yan / Somnath Mukherjee / Kaleeckal G Harikumar / Timothy S Strutzenberg / X Edward Zhou / Kelly Suino-Powell / Ting-Hai Xu / Ryan D Sheldon / Jared Lamp / Joseph S Brunzelle / Katarzyna ... 著者 : Yan Yan / Somnath Mukherjee / Kaleeckal G Harikumar / Timothy S Strutzenberg / X Edward Zhou / Kelly Suino-Powell / Ting-Hai Xu / Ryan D Sheldon / Jared Lamp / Joseph S Brunzelle / Katarzyna Radziwon / Abigail Ellis / Scott J Novick / Irving E Vega / Russell G Jones / Laurence J Miller / H Eric Xu / Patrick R Griffin / Anthony A Kossiakoff / Karsten Melcher / 要旨 : Adenosine monophosphate (AMP)-activated protein kinase (AMPK) regulates metabolism in response to the cellular energy states. Under energy stress, AMP stabilizes the active AMPK conformation, in ... Adenosine monophosphate (AMP)-activated protein kinase (AMPK) regulates metabolism in response to the cellular energy states. Under energy stress, AMP stabilizes the active AMPK conformation, in which the kinase activation loop (AL) is protected from protein phosphatases, thus keeping the AL in its active, phosphorylated state. At low AMP:ATP (adenosine triphosphate) ratios, ATP inhibits AMPK by increasing AL dynamics and accessibility. We developed conformation-specific antibodies to trap ATP-bound AMPK in a fully inactive, dynamic state and determined its structure at 3.5-angstrom resolution using cryo-electron microscopy. A 180° rotation and 100-angstrom displacement of the kinase domain fully exposes the AL. On the basis of the structure and supporting biophysical data, we propose a multistep mechanism explaining how adenine nucleotides and pharmacological agonists modulate AMPK activity by altering AL phosphorylation and accessibility. 履歴 登録 2020年7月20日 - ヘッダ(付随情報) 公開 2021年7月21日 - マップ公開 2021年7月21日 - 更新 2021年12月15日 - 現状 2021年12月15日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) / synthetic construct (人工物) /
Homo sapiens (ヒト) / synthetic construct (人工物) / 
 データ登録者
データ登録者 米国, 2件
米国, 2件  引用
引用 ジャーナル: Science / 年: 2021
ジャーナル: Science / 年: 2021

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_22337.map.gz
emd_22337.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-22337-v30.xml
emd-22337-v30.xml emd-22337.xml
emd-22337.xml EMDBヘッダ
EMDBヘッダ emd_22337_fsc.xml
emd_22337_fsc.xml FSCデータファイル
FSCデータファイル emd_22337.png
emd_22337.png http://ftp.pdbj.org/pub/emdb/structures/EMD-22337
http://ftp.pdbj.org/pub/emdb/structures/EMD-22337 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22337
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22337 emd_22337_validation.pdf.gz
emd_22337_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_22337_full_validation.pdf.gz
emd_22337_full_validation.pdf.gz emd_22337_validation.xml.gz
emd_22337_validation.xml.gz emd_22337_validation.cif.gz
emd_22337_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22337
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22337 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22337
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22337 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_22337.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_22337.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー