+検索条件
-Structure paper
| タイトル | Structural mobility tunes signalling of the GluA1 AMPA glutamate receptor. |
|---|---|
| ジャーナル・号・ページ | Nature, Vol. 621, Issue 7980, Page 877-882, Year 2023 |
| 掲載日 | 2023年9月13日 |
 著者 著者 | Danyang Zhang / Josip Ivica / James M Krieger / Hinze Ho / Keitaro Yamashita / Imogen Stockwell / Rozbeh Baradaran / Ondrej Cais / Ingo H Greger /   |
| PubMed 要旨 | AMPA glutamate receptors (AMPARs), the primary mediators of excitatory neurotransmission in the brain, are either GluA2 subunit-containing and thus Ca-impermeable, or GluA2-lacking and Ca-permeable. ...AMPA glutamate receptors (AMPARs), the primary mediators of excitatory neurotransmission in the brain, are either GluA2 subunit-containing and thus Ca-impermeable, or GluA2-lacking and Ca-permeable. Despite their prominent expression throughout interneurons and glia, their role in long-term potentiation and their involvement in a range of neuropathologies, structural information for GluA2-lacking receptors is currently absent. Here we determine and characterize cryo-electron microscopy structures of the GluA1 homotetramer, fully occupied with TARPγ3 auxiliary subunits (GluA1/γ3). The gating core of both resting and open-state GluA1/γ3 closely resembles GluA2-containing receptors. However, the sequence-diverse N-terminal domains (NTDs) give rise to a highly mobile assembly, enabling domain swapping and subunit re-alignments in the ligand-binding domain tier that are pronounced in desensitized states. These transitions underlie the unique kinetic properties of GluA1. A GluA2 mutant (F231A) increasing NTD dynamics phenocopies this behaviour, and exhibits reduced synaptic responses, reflecting the anchoring function of the AMPAR NTD at the synapse. Together, this work underscores how the subunit-diverse NTDs determine subunit arrangement, gating properties and ultimately synaptic signalling efficiency among AMPAR subtypes. |
 リンク リンク |  Nature / Nature /  PubMed:37704721 / PubMed:37704721 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.64 - 3.77 Å |
| 構造データ | EMDB-16379, PDB-8c1p: EMDB-16380, PDB-8c1q: EMDB-16381, PDB-8c1r: EMDB-16382, PDB-8c1s: EMDB-16390, PDB-8c2h: EMDB-16391, PDB-8c2i: EMDB-17392, PDB-8p3q: EMDB-17393, PDB-8p3s: EMDB-17394, PDB-8p3t: EMDB-17395, PDB-8p3u: EMDB-17396, PDB-8p3v: EMDB-17397, PDB-8p3w: EMDB-17398, PDB-8p3x: EMDB-17399, PDB-8p3y: EMDB-17400, PDB-8p3z: EMDB-17692, PDB-8piv: |
| 化合物 |  ChemComp-PLM:  ChemComp-OLC:  ChemComp-GLU:  ChemComp-POV:  ChemComp-HOH: 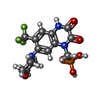 ChemComp-ZK1: |
| 由来 |
|
 キーワード キーワード | MEMBRANE PROTEIN / AMPAR / ion channels / neurotransmission / AMPA-type glutamate neurotransmitter receptor / auxiliary subunit complex / agonist / desensitized |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について