-検索条件
-検索結果
検索 (著者・登録者: del & vas & m)の結果全44件を表示しています
EMDB-40601: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa
EMDB-40625: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa
EMDB-40626: 
cytochrome bc1 complex from Pseudomonas aeruginosa

EMDB-40627: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3)
EMDB-40637: 
cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined bc1 monomer with Rieske head domain in b state)
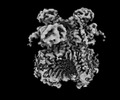
EMDB-40638: 
cytochrome bc1 complex from Pseudomonas aeruginosa (Locally refined bc1 monomer with Rieske head domain in c state)

EMDB-40643: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3 with CcoP1 isoform)
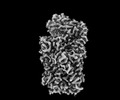
EMDB-40645: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa (Locally refined cytochrome cbb3 with CcoP2 isoform)

PDB-8smr: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa

PDB-8snh: 
cytochrome bc1-cbb3 supercomplex from Pseudomonas aeruginosa

EMDB-15713: 
Structure of Complement C5 in Complex with small molecule inhibitor and CVF

PDB-8ayh: 
Structure of Complement C5 in Complex with small molecule inhibitor and CVF

EMDB-26936: 
Complex of Plasmodium falciparum circumsporozoite protein with 850 Fab

PDB-7v05: 
Complex of Plasmodium falciparum circumsporozoite protein with 850 Fab

PDB-7t3h: 
MicroED structure of Dynobactin

EMDB-14633: 
PucA-LH2 complex from Rps. palustris

PDB-7zcu: 
PucA-LH2 complex from Rps. palustris

EMDB-14650: 
PucB-LH2 complex from Rps. palustris

EMDB-14682: 
PucD-LH2 complex from Rps. palustris

EMDB-14685: 
PucE-LH2 complex from Rps. palustris

PDB-7zdi: 
PucB-LH2 complex from Rps. palustris

PDB-7ze3: 
PucD-LH2 complex from Rps. palustris

PDB-7ze8: 
PucE-LH2 complex from Rps. palustris

EMDB-14242: 
E. coli BAM complex (BamABCDE) bound to dynobactin A

PDB-7r1w: 
E. coli BAM complex (BamABCDE) bound to dynobactin A
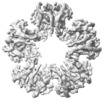
EMDB-23046: 
Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (decamer)

EMDB-23047: 
Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (dodecamer)

PDB-7kvc: 
Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (decamer)

PDB-7kvd: 
Cryo-EM structure of Mal de Rio Cuarto virus P9-1 viroplasm protein (dodecamer)

EMDB-11953: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Composite Map

EMDB-11954: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 2)

EMDB-14810: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Consensus Map

EMDB-14811: 
SARS-CoV-2 S 2P trimer in complex with monovalent DARPin R2 (State 1) - Focused Refinement

EMDB-4995: 
Subtomogram average of a part of the rabies lyssavirus ribonucleoprotein

EMDB-0176: 
Cryo-EM structure of a 70S Bacillus subtilis ribosome translating the ErmD leader peptide in complex with telithromycin

EMDB-0177: 
Cryo-EM structure of the ABCF protein VmlR bound to the Bacillus subtilis ribosome

PDB-6ha1: 
Cryo-EM structure of a 70S Bacillus subtilis ribosome translating the ErmD leader peptide in complex with telithromycin

PDB-6ha8: 
Cryo-EM structure of the ABCF protein VmlR bound to the Bacillus subtilis ribosome

EMDB-3357: 
electron density map of murine leukaemia virus envelope glycoprotein tagged in the proline rich region with YFP as reconstructed by subtomogram averaging on viruses produced in DFJ8 cells

EMDB-3363: 
electron density map of murine leukaemia virus envelope glycoprotein tagged in the proline rich region with YFP as reconstructed by subtomogram averaging on murine leukemia virus virus like particles (3 plasmid system) produced in COS1 cells

EMDB-3365: 
electron density map of murine leukaemia virus envelope glycoprotein as reconstructed by subtomogram averaging on murine leukemia virus virus like particles (3 plasmid system) produced in COS1 cells

EMDB-3373: 
electron density map of murine leukaemia virus envelope glycoprotein as reconstructed by subtomogram averaging applying a mask on 1 protomer on murine leukemia virus particles and virus like particles and applying 3fold symmetry to the single protomer afterwards

EMDB-4001: 
Cryo-EM structure of stringent response factor RelA bound to ErmCL-stalled ribosome complex

PDB-5l3p: 
Cryo-EM structure of stringent response factor RelA bound to ErmCL-stalled ribosome complex
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
