-検索条件
-検索結果
検索 (著者・登録者: zhang & xz)の結果67件中、1から50件目までを表示しています
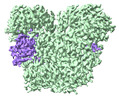
EMDB-36980: 
Cryo-EM structure of DSR2-TTP

EMDB-36982: 
Cryo-EM structure of DSR2-DSAD1 state 2

EMDB-37272: 
Cryo-EM structure of DSR2-DSAD1 state 1
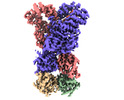
EMDB-37603: 
Cryo-EM structure of DSR2-DSAD1

EMDB-38421: 
Cryo-EM structure of tail tube protein
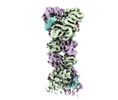
EMDB-37497: 
Cryo-EM structure of DSR2-TTP

EMDB-36343: 
The cryo-EM structure of Parvovirus milled by 30 keV gallium FIB at 3.09 Angstrom resolution.

EMDB-36346: 
The cryo-EM structure of 10-20nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 4.05 Angstrom resolution.

EMDB-36347: 
The cryo-EM structure of 20-30nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 3.88 Angstrom resolution.

EMDB-36348: 
The cryo-EM structure of 30-40 nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 3.82 Angstrom resolution.

EMDB-36349: 
The cryo-EM structure of 40-50 nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 3.62 Angstrom resolution.

EMDB-36350: 
The cryo-EM structure of 50-60 nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 3.60 Angstrom resolution.

EMDB-36351: 
The cryo-EM structure of 60-70 nm group Parvovirus from lamellae surface milled by 30 keV Ga+ FIB at 3.57 Angstrom resolution.
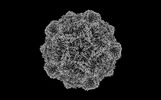
EMDB-36352: 
The cryo-EM structure of Parvovirus milled by 8 keV gallium FIB at 3.11 Angstrom resolution.

EMDB-36355: 
The cryo-EM structure of 10-20 nm group Parvovirus from lamellae surface milled by 8 keV Ga+ FIB at 3.88 Angstrom resolution.

EMDB-36356: 
The cryo-EM structure of 20-30 nm group Parvovirus from lamellae surface milled by 8 keV Ga+ FIB at 3.70 Angstrom resolution.

EMDB-36357: 
The cryo-EM structure of 30-40 nm group Parvovirus from lamellae surface milled by 8 keV Ga+ FIB at 3.60 Angstrom resolution.

EMDB-36358: 
The cryo-EM structure of 40-50 nm group Parvovirus from lamellae surface milled by 8 keV Ga+ FIB at 3.54 Angstrom resolution.

EMDB-36359: 
The cryo-EM structure of 50-60 nm group Parvovirus from lamellae surface milled by 8 keV Ga+ FIB at 3.57 Angstrom resolution.

EMDB-36363: 
The cryo-EM structure of ribosome from yeast lamellae milled by 8 keV Ga+ FIB at 3.78 Angstrom resolution.

EMDB-35240: 
Cryo-EM structure of TIR-APAZ/Ago-gRNA-DNA complex

EMDB-35241: 
Cryo-EM structure of TIR-APAZ/Ago-gRNA complex

EMDB-35592: 
Cryo-EM structure of the target ssDNA-bound SIR2-APAZ/Ago-gRNA quaternary complex

EMDB-34227: 
Cryo-EM structure of Singapore Grouper Iridovirus capsid block 1

EMDB-34229: 
Cryo-EM structure of Singapore Grouper Iridovirus capsid block 4

EMDB-34230: 
Cryo-EM structure of Singapore Grouper Iridovirus capsid block 2

EMDB-34235: 
Cryo-EM structure of Singapore Grouper Iridovirus capsid block 3

EMDB-34236: 
Cryo-EM structure of Singapore Grouper Iridovirus capsid block 5
EMDB-34251: 
Singapore Grouper Iridovirus

EMDB-34815: 
One asymmetric unit of Singapore grouper iridovirus capsid

EMDB-31803: 
LolCDE with bound RcsF in nanodiscs

EMDB-32611: 
Structure of Coxsackievirus A10 with Hybrid electron counting at 200 kV

EMDB-32600: 
Structure of Coxsackievirus A10 for critical dose measurement at 120 kV

EMDB-32601: 
Structure of Coxsackievirus A10 for critical dose measurement at 160 kV

EMDB-32602: 
Structure of Coxsackievirus A10 for critical dose measurement at 300 kV

EMDB-32603: 
Structure of Coxsackievirus A10 with Hybrid electron counting at 120 kV

EMDB-32604: 
Structure of Coxsackievirus A10 with MCF electron counting at 120 kV

EMDB-32605: 
Structure of Coxsackievirus A10 with WPF electron counting at 120 kV

EMDB-32606: 
Structure of Coxsackievirus A10 with MCF electron counting and large-sized clusters at 120 kV

EMDB-32607: 
Structure of Coxsackievirus A10 with PVF electron counting and large-sized clusters at 120 kV

EMDB-32608: 
Structure of Coxsackievirus A10 with PCA electron counting and large-sized clusters at 120 kV

EMDB-32609: 
Structure of Coxsackievirus A10 with MCF electron counting at 200 kV

EMDB-32610: 
Structure of apo-ferritin with PCA electron counting at 300 kV

EMDB-32612: 
Structure of apo-ferritin with Hybrid electron counting at 120 kV

EMDB-32613: 
Structure of apo-ferritin with MCF electron counting at 120 kV

EMDB-25673: 
Prepore structure of pore-forming toxin Epx1

EMDB-31482: 
Cryo-EM structure of the human TACAN channel in a closed state

EMDB-30646: 
Structure of Calcium-Sensing Receptor in an inactive state

EMDB-9994: 
Structure of cyanobacterial photosystem I-IsiA-flavodoxin supercomplex

EMDB-9995: 
Structure of cyanobacterial photosystem I-IsiA supercomplex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
