-検索条件
-検索結果
検索 (著者・登録者: toropova & k)の結果全48件を表示しています

EMDB-19132: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

EMDB-19133: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

PDB-8rgg: 
Structure of dynein-2 intermediate chain DYNC2I2 (WDR34) in complex with dynein-2 heavy chain DYNC2H1.

PDB-8rgh: 
Structure of dynein-2 intermediate chain DYNC2I1 (WDR60) in complex with the dynein-2 heavy chain DYNC2H1.

EMDB-15954: 
Structure of the IFT-A complex; IFT-A2 module

PDB-8bbe: 
Structure of the IFT-A complex; IFT-A2 module

PDB-8bbg: 
Structure of the IFT-A complex; anterograde IFT-A train model

EMDB-15955: 
Structure of the IFT-A complex; IFT-A1 module

PDB-8bbf: 
Structure of the IFT-A complex; IFT-A1 module

PDB-6sc2: 
Structure of the dynein-2 complex; IFT-train bound model

EMDB-4917: 
Structure of the dynein-2 complex; motor domains

EMDB-4918: 
Structure of the dynein-2 complex; tail domain

PDB-6rla: 
Structure of the dynein-2 complex; motor domains

PDB-6rlb: 
Structure of the dynein-2 complex; tail domain

EMDB-8484: 
Bacteriophage G

EMDB-8485: 
Bacteriophage 121Q

EMDB-8486: 
Bacillus phage PBS1

EMDB-8487: 
Bacteriophage N3

EMDB-8488: 
Bacteriophage PAU

EMDB-8489: 
Bacteriophage Bellamy

EMDB-6008: 
Dynein motor domain in complex with Lis1 in the absence of nucleotide
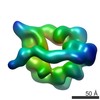
EMDB-6013: 
Dynein motor domain in the absence of nucleotide

EMDB-6014: 
Dynein motor domain in the presence of ADP, with linker at position 2
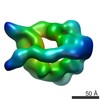
EMDB-6015: 
Dynein motor domain in the presence of ADP, with linker at position 1
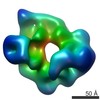
EMDB-6016: 
Dynein motor domain in complex with Lis1 in the presence of ATP and Vi

EMDB-6017: 
Dynein motor domain with truncated linker in complex with Lis1 in the absence of nucleotide

EMDB-5650: 
Pseudorabies virus C-capsid from cryo-electron microscopy

EMDB-5652: 
Pseudorabies virus B-capsid from cryo-electron microscopy

EMDB-5654: 
Pseudorabies virus A-capsid from cryo-electron microscopy

EMDB-5655: 
Pseudorabies virus virion capsid from cryo-electron microscopy

EMDB-5656: 
Pseudorabies GS-2168 B-capsid from cryo-electron microscopy

EMDB-5657: 
Structure of the pseudorabies virus capsid: comparison with herpes simplex virus type 1 and differential binding of essential minor proteins

EMDB-5659: 
Cryo-electron microscopy of herpesvirus simplex type 1 C-capsid

EMDB-5660: 
Cryo-electron microscopy of herpesvirus simplex type 1 B-capsid

EMDB-5661: 
Cryo-electron microscopy of herpesvirus simplex type 1 A-capsid

EMDB-1902: 
The HSV-1 UL17 protein is the second constituent of the capsid vertex specific component (CVSC) required for DNA packaging and retention.

EMDB-1903: 
The HSV-1 UL17 protein is the second constituent of the capsid vertex specific component (CVSC) required for DNA packaging and retention.

EMDB-1905: 
Residues of the UL25 Protein of Herpes Simplex Virus That Are Required for Its Stable Interaction with Capsids.

EMDB-1879: 
Mutually-induced conformational switching of RNA and coat protein underpins efficient assembly of a viral capsid.

EMDB-1880: 
Mutually-induced conformational switching of RNA and coat protein underpins efficient assembly of a viral capsid.

EMDB-1881: 
Mutually-induced conformational switching of RNA and coat protein underpins efficient assembly of a viral capsid.

EMDB-1882: 
Mutually-induced conformational switching of RNA and coat protein underpins efficient assembly of a viral capsid.
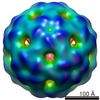
EMDB-1860: 
Visualising an RNA genome poised for release from its receptor complex.

EMDB-1859: 
Visualising a viral RNA genome poised for release from its receptor complex.

EMDB-1861: 
Visualising an RNA genome poised for release from its receptor complex.

EMDB-1431: 
The three-dimensional structure of genomic RNA in bacteriophage MS2: implications for assembly.

EMDB-1432: 
The three-dimensional structure of genomic RNA in bacteriophage MS2: implications for assembly.

EMDB-1433: 
The three-dimensional structure of genomic RNA in bacteriophage MS2: implications for assembly.
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
