-検索条件
-検索結果
検索 (著者・登録者: terry & le)の結果全45件を表示しています

EMDB-29637: 
Dehosphorylated, ATP-bound human cystic fibrosis transmembrane conductance regulator (CFTR)

PDB-8fzq: 
Dehosphorylated, ATP-bound human cystic fibrosis transmembrane conductance regulator (CFTR)

EMDB-33241: 
Cryo-EM Structure of Human Niacin Receptor HCA2-Gi protein complex

PDB-7xk2: 
Cryo-EM Structure of Human Niacin Receptor HCA2-Gi protein complex
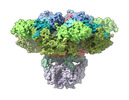
EMDB-27982: 
Structure of the full-length IP3R1 channel determined at high Ca2+
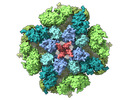
EMDB-27983: 
Structure of the full-length IP3R1 channel determined in the presence of Calcium/IP3/ATP

PDB-8eaq: 
Structure of the full-length IP3R1 channel determined at high Ca2+

PDB-8ear: 
Structure of the full-length IP3R1 channel determined in the presence of Calcium/IP3/ATP
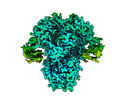
EMDB-26727: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation
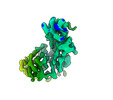
EMDB-26729: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)
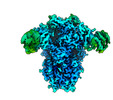
EMDB-26730: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation
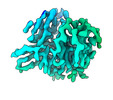
EMDB-26731: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

PDB-7us6: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the proximal conformation

PDB-7us9: 
CCoV-HuPn-2018 S in the proximal conformation (local refinement of domain 0)

PDB-7usa: 
Structure of the human coronavirus CCoV-HuPn-2018 spike glycoprotein with domain 0 in the swung out conformation

PDB-7usb: 
CCoV-HuPn-2018 S in the swung out conformation (local refinement of domain 0)

EMDB-23494: 
Cryo-EM of the SLFN12-PDE3A complex: PDE3A body refinement

EMDB-23495: 
Cryo-EM of the SLFN12-PDE3A complex: Consensus subset model

EMDB-23496: 
Cryo-EM of the SLFN12-PDE3A complex: SLFN12 body refinement

PDB-7lrc: 
Cryo-EM of the SLFN12-PDE3A complex: PDE3A body refinement

PDB-7lrd: 
Cryo-EM of the SLFN12-PDE3A complex: Consensus subset model

PDB-7lre: 
Cryo-EM of the SLFN12-PDE3A complex: SLFN12 body refinement

EMDB-20121: 
Structure of trans-translation inhibitor bound to E. coli 70S ribosome with P site tRNA

PDB-6om6: 
Structure of trans-translation inhibitor bound to E. coli 70S ribosome with P site tRNA

EMDB-0173: 
Complex of foot-and-mouth-disease virus (type O1 M) with the Fab of antibody D9. The virus is at 3.5 Ang resolution, the Fab is flexibly attached and at much lower resolution.

PDB-5ler: 
Structure of the bacterial sex F pilus (13.2 Angstrom rise)

PDB-5lfb: 
Structure of the bacterial sex F pilus (12.5 Angstrom rise)

PDB-5leg: 
Structure of the bacterial sex F pilus (pED208)

EMDB-4042: 
Cryo-EM structure of the bacterial sex F pilus (pED208)

EMDB-4044: 
Cryo-EM structure of the bacterial sex F pilus (13.2 Angstrom rise)

EMDB-4046: 
Cryo-EM structure of the bacterial sex F pilus (12.5 Angstrom rise)

EMDB-4092: 
Tomographic reconstruction of ex vivo mammalian prions

EMDB-3129: 
Structure-based energetics of protein interfaces guide FMDV vaccine design

EMDB-3130: 
Structure-based energetics of protein interfaces guide FMDV vaccine design

PDB-5ac9: 
Structure-based energetics of protein interfaces guide Foot-and-Mouth disease virus vaccine design

PDB-5aca: 
Structure-based energetics of protein interfaces guide Foot-and-Mouth disease virus vaccine design

EMDB-2433: 
Amyloid-beta nanotube

EMDB-5041: 
Ribosome structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5042: 
Lumazine synthase structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5043: 
GroEL structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5044: 
RNA polymerase structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5045: 
Phosphoenolpyruvate synthase structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5046: 
Putative protein structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)

EMDB-5047: 
Inosine-5-monophosphate dehydrogenase structure : Structural survey of large protein complexes in Desulfovibrio vulgaris Hildenborough (DvH)
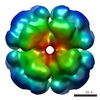
EMDB-1319: 
Octomeric pyruvate-ferredoxin oxidoreductase from Desulfovibrio vulgaris.
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
