-検索条件
-検索結果
検索 (著者・登録者: sui & j)の結果322件中、1から50件目までを表示しています

EMDB-36635: 
Structure of arginine oxidase from Pseudomonas sp. TRU 7192

EMDB-42897: 
LapB cytoplasmic domain in complex with LpxC

PDB-8v24: 
LapB cytoplasmic domain in complex with LpxC

EMDB-37984: 
Structure of nucleosome-bound SRCAP-C in the apo state

EMDB-37988: 
Structure of nucleosome-bound SRCAP-C in the ADP-BeFx-bound state

EMDB-37990: 
Structure of nucleosome-bound SRCAP-C in the ADP-bound state

PDB-8x15: 
Structure of nucleosome-bound SRCAP-C in the apo state

PDB-8x19: 
Structure of nucleosome-bound SRCAP-C in the ADP-BeFx-bound state

PDB-8x1c: 
Structure of nucleosome-bound SRCAP-C in the ADP-bound state
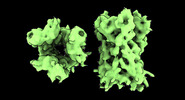
EMDB-17704: 
Subtomogram average of Vaccinia A10 trimer with open center from in vitro cores
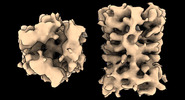
EMDB-17708: 
Subtomogram average of Vaccinia A10 trimer with tight center from in vitro cores

EMDB-17753: 
Subtomogram average of Vaccinia A10 trimer from in situ cores

EMDB-36020: 
Cryo-EM structure of the Arabidopsis thaliana photosystem I(PSI-LHCII-ST2)

EMDB-36021: 
Cryo-EM structure of the Arabidopsis thaliana photosystem I(PSI-LHCII-ST2)

EMDB-36022: 
LHCI of PSI-LHCII from Arabidopsis thaliana(state2)

EMDB-36023: 
LHCII of PSI-LHCII from Arabidopsis thaliana(state2)

EMDB-36026: 
Cryo-EM structure of the Arabidopsis thaliana photosystem I in state 2(PSI-ST2)

EMDB-36032: 
LHCI of PSI from Arabidopsis thaliana in state 2 (PSI-ST2)

EMDB-36033: 
Cryo-EM structure of the Arabidopsis thaliana PSI in state 1 (PSI-ST1)

EMDB-36035: 
LHCI of PSI from Arabidopsis thaliana in state 1 (PSI-ST1)

EMDB-36036: 
Coordinates of Cryo-EM structure of the Arabidopsis thaliana PSI in state 1 (PSI-ST1)

EMDB-36037: 
Coordinates of Cryo-EM structure of the Arabidopsis thaliana PSI in state 2 (PSI-ST2)

PDB-8j6z: 
Cryo-EM structure of the Arabidopsis thaliana photosystem I(PSI-LHCII-ST2)

PDB-8j7a: 
Coordinates of Cryo-EM structure of the Arabidopsis thaliana PSI in state 1 (PSI-ST1)

PDB-8j7b: 
Coordinates of Cryo-EM structure of the Arabidopsis thaliana PSI in state 2 (PSI-ST2)

EMDB-34534: 
Un-sharpened map of phycobilisome from Porphyridium purpureum under Middle light

EMDB-34529: 
Un-sharpened map of phycobilisome from Porphyridium purpureum under low light

EMDB-28552: 
Human Membrane-bound O-acyltransferase 7

PDB-8erc: 
Human Membrane-bound O-acyltransferase 7

EMDB-34530: 
Membrane protein A

EMDB-34531: 
Membrane protein B

EMDB-35713: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

PDB-8h86: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 in lipid nanodisc

PDB-8h87: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR2 in lipid nanodisc

PDB-8iu0: 
Cryo-EM structure of the potassium-selective channelrhodopsin HcKCR1 H225F mutant in lipid nanodisc

EMDB-17198: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (prior to enrichment of particles containing TIM-1/TIPN-1)

EMDB-17199: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement DNSN-1 homodimer)

EMDB-17200: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement GINS/CDC-45/DNSN-1 N-terminus)

EMDB-17201: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA+/ssDNA)

EMDB-17202: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 NTDs)

EMDB-17203: 
Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement TIM-1/TIPN-1/MCM-2-7 NTD/dsDNA)

EMDB-17204: 
Cryo-EM structure of CMG helicase bound to TIM-1/TIPN-1 and homodimeric DNSN-1 on fork DNA (Caenorhabditis elegans)

PDB-8ouw: 
Cryo-EM structure of CMG helicase bound to TIM-1/TIPN-1 and homodimeric DNSN-1 on fork DNA (Caenorhabditis elegans)

PDB-8jng: 
Methanesulfonate monooxygenase ferredoxin subunit of PBS-PSII-PSI-LHCs from Porphyridium purpureum.

PDB-8jnl: 
Psb34 from red algal Porphyridium purpureum.

PDB-8jnm: 
PsbW from red algal Porphyridium purpureum.

PDB-8jnn: 
linker protein LPP1 from red algal Porphyridium purpureum.

EMDB-33669: 
In situ double-PBS-PSII-PSI-LHCs megacomplex from red alga Porphyridium purpureum.

EMDB-40983: 
Negative stain EM assembly of MYC, JAZ, and NINJA complex

PDB-8t2i: 
Negative stain EM assembly of MYC, JAZ, and NINJA complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
