[English] 日本語
 Yorodumi
Yorodumi- EMDB-17201: Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA+/ssDNA) | |||||||||
 Map data Map data | Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA /ssDNA) | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Replisome / DONSON / DNA replication / Initiation / REPLICATION | |||||||||
| Biological species |  | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.17 Å | |||||||||
 Authors Authors | Jenkyn-Bedford M / Yeeles JTP | |||||||||
| Funding support |  United Kingdom, 2 items United Kingdom, 2 items
| |||||||||
 Citation Citation |  Journal: Science / Year: 2023 Journal: Science / Year: 2023Title: DNSN-1 recruits GINS for CMG helicase assembly during DNA replication initiation in . Authors: Yisui Xia / Remi Sonneville / Michael Jenkyn-Bedford / Liqin Ji / Constance Alabert / Ye Hong / Joseph T P Yeeles / Karim P M Labib /   Abstract: Assembly of the CMG (CDC-45-MCM-2-7-GINS) helicase is the key regulated step during eukaryotic DNA replication initiation. Until now, it was unclear whether metazoa require additional factors that ...Assembly of the CMG (CDC-45-MCM-2-7-GINS) helicase is the key regulated step during eukaryotic DNA replication initiation. Until now, it was unclear whether metazoa require additional factors that are not present in yeast. In this work, we show that DNSN-1, the ortholog of human DONSON, functions during helicase assembly in a complex with MUS-101/TOPBP1. DNSN-1 is required to recruit the GINS complex to chromatin, and a cryo-electron microscopy structure indicates that DNSN-1 positions GINS on the MCM-2-7 helicase motor (comprising the six MCM-2 to MCM-7 proteins), by direct binding of DNSN-1 to GINS and MCM-3, using interfaces that we show are important for initiation and essential for viability. These findings identify DNSN-1 as a missing link in our understanding of DNA replication initiation, suggesting that initiation defects underlie the human disease syndrome that results from DONSON mutations. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_17201.map.gz emd_17201.map.gz | 70 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-17201-v30.xml emd-17201-v30.xml emd-17201.xml emd-17201.xml | 16.6 KB 16.6 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_17201.png emd_17201.png | 112 KB | ||
| Filedesc metadata |  emd-17201.cif.gz emd-17201.cif.gz | 4.4 KB | ||
| Others |  emd_17201_half_map_1.map.gz emd_17201_half_map_1.map.gz emd_17201_half_map_2.map.gz emd_17201_half_map_2.map.gz | 87.5 MB 87.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17201 http://ftp.pdbj.org/pub/emdb/structures/EMD-17201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17201 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17201 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_17201.map.gz / Format: CCP4 / Size: 120.4 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_17201.map.gz / Format: CCP4 / Size: 120.4 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA /ssDNA) | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.295 Å | ||||||||||||||||||||||||||||||||||||
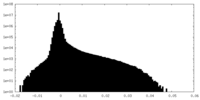
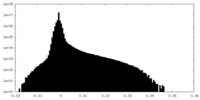
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: Half-map 1. Cryo-EM density map of the C....
| File | emd_17201_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half-map 1. Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA /ssDNA) | ||||||||||||
| Projections & Slices |
| ||||||||||||
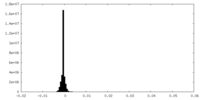
| Density Histograms |
-Half map: Half-map 2. Cryo-EM density map of the C....
| File | emd_17201_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half-map 2. Cryo-EM density map of the C. elegans complex CMG helicase/TIM-1/TIPN-1/DNSN-1 dimer on fork DNA (Multi-body Refinement MCM-2-7 AAA /ssDNA) | ||||||||||||
| Projections & Slices |
| ||||||||||||
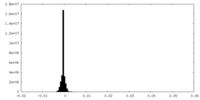
| Density Histograms |
- Sample components
Sample components
-Entire : In vitro reconstituted complex of Caenorhabditis elegans CMG heli...
| Entire | Name: In vitro reconstituted complex of Caenorhabditis elegans CMG helicase bound by TIM-1/TIPN-1 and a homodimer of DNSN-1, on a synthetic forked DNA substrate |
|---|---|
| Components |
|
-Supramolecule #1: In vitro reconstituted complex of Caenorhabditis elegans CMG heli...
| Supramolecule | Name: In vitro reconstituted complex of Caenorhabditis elegans CMG helicase bound by TIM-1/TIPN-1 and a homodimer of DNSN-1, on a synthetic forked DNA substrate type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#16 Details: All Caenorhabditis elegans proteins recombinantly expressed in either Saccharomyces cerevisiae (CMG helicase, TIM-1/TIPN-1) or Escherichia coli Rosetta (DE3) Competent cells (DNSN-1) |
|---|---|
| Source (natural) | Organism:  |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.5 |
|---|---|
| Grid | Model: Quantifoil R2/2 / Material: COPPER / Mesh: 400 / Support film - Material: CARBON / Support film - topology: CONTINUOUS / Support film - Film thickness: 2 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 5 sec. / Details: PELCO easyGlow. 15 mA |
| Vitrification | Cryogen name: ETHANE / Details: Manual plunger. |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 BIOQUANTUM (6k x 4k) / Number real images: 10825 / Average exposure time: 1.7 sec. / Average electron dose: 40.1 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.5 µm / Nominal defocus min: 1.0 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)