-検索条件
-検索結果
検索 (著者・登録者: speck & c)の結果52件中、1から50件目までを表示しています

EMDB-13619: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III (composite map)

EMDB-13620: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, state I (composite map)

EMDB-13621: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III (B1 map)

EMDB-13622: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III (B2 map)

EMDB-13623: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III (B3 map)

EMDB-13624: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III (3D auto-refined map)

EMDB-13629: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state II (composite map)

EMDB-13631: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state II (B1 map)

EMDB-13635: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state II (B2 map)

EMDB-13640: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state II (3D auto-refined map)

EMDB-13644: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state I (3D auto-refined map)

EMDB-13645: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, state I (B1 map)

EMDB-13646: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, state I (B2 map)

EMDB-13647: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, state I (3D auto-refined map)

EMDB-13648: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state (3D auto-refined map)

EMDB-13649: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state A (binned 3D auto-refined map)

EMDB-13650: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state B (binned 3D auto-refined map)

EMDB-13651: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state C (binned 3D auto-refined map)

EMDB-13652: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state D (binned 3D auto-refined map)

EMDB-13653: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state E (binned 3D auto-refined map)

EMDB-13655: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, swiveled state F (binned 3D auto-refined map)

EMDB-13656: 
Structure of double-stranded DNA-bound MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATP (3D auto-refined map)

EMDB-13657: 
Structure of double-stranded DNA-bound MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATP (B1 map)

EMDB-13658: 
Structure of double-stranded DNA-bound MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATP (B2 map)

EMDB-13659: 
Structure of double-stranded DNA-bound MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATP (B3 map)

PDB-7pt6: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ATPgS, state III

PDB-7pt7: 
Structure of MCM2-7 DH complexed with Cdc7-Dbf4 in the presence of ADP:BeF3, state I

EMDB-13453: 
Cryo-EM structure of the agonist setmelanotide bound to the active melanocortin-4 receptor (MC4R) in complex with the heterotrimeric Gs protein at 2.6 A resolution.

EMDB-13454: 
Active Melanocortin-4 receptor (MC4R)- Gs protein complex bound to agonist NDP-alpha-MSH at 2.86 A resolution.

PDB-7piu: 
Cryo-EM structure of the agonist setmelanotide bound to the active melanocortin-4 receptor (MC4R) in complex with the heterotrimeric Gs protein at 2.6 A resolution.

PDB-7piv: 
Active Melanocortin-4 receptor (MC4R)- Gs protein complex bound to agonist NDP-alpha-MSH at 2.86 A resolution.

EMDB-23755: 
CryoEM map of the structure of the S. cerevisiae origin recognition complex bound to the replication initiator Cdc6 and the ARS1 origin DNA.

EMDB-23818: 
Cryo-EM 3D map of the S. cerevisiae origin recognition complex bound to Cdc6 and ARS1 origin DNA

PDB-7mca: 
Structure of the S. cerevisiae origin recognition complex bound to the replication initiator Cdc6 and the ARS1 origin DNA.

EMDB-21662: 
Cryo-EM map of semi-attached mutant OCCM-DNA complex (ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation)

EMDB-21664: 
Cryo-EM map of mutant Mcm2-7 hexamer with Mcm6 WHD truncation

EMDB-21665: 
Cryo-EM map of pre-insertion mutant OCCM-DNA complex (ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation)

EMDB-21666: 
Cryo-EM map of the mutant OCCM (ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation) loaded on DNA at 10.5 A resolution

PDB-6wgc: 
Atomic model of semi-attached mutant OCCM-DNA complex (ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation)

PDB-6wgf: 
Atomic model of mutant Mcm2-7 hexamer with Mcm6 WHD truncation

PDB-6wgg: 
Atomic model of pre-insertion mutant OCCM-DNA complex(ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation)

PDB-6wgi: 
Atomic model of the mutant OCCM (ORC-Cdc6-Cdt1-Mcm2-7 with Mcm6 WHD truncation) loaded on DNA at 10.5 A resolution

EMDB-9400: 
Cryo-EM structure of Mcm2-7 double hexamer on dsDNA

PDB-5bk4: 
Cryo-EM structure of Mcm2-7 double hexamer on dsDNA

PDB-5v8f: 
Structural basis of MCM2-7 replicative helicase loading by ORC-Cdc6 and Cdt1

EMDB-8540: 
Structural basis of MCM2-7 replicative helicase loading by ORC-Cdc6 and Cdt1

EMDB-5857: 
Architectures of Mcm2-7 Double Hexamer Assembly Intermediates Reveal Helicase Loading Mechanism

EMDB-5625: 
Architecture of a helicase loading intermediate containing ORC-Cdc6-Cdt1-MCM2-7 on DNA reveals similarity to DNA polymerase clamp loading complexes

EMDB-5381: 
Cdc6-induced Conformational Changes in ORC Bound To Origin DNA Revealed by Cryo-Electron Microscopy. This map may be mirrored (based on comparison to EMD-5625).
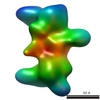
EMDB-5013: 
A 3D EM map of the subcomplex Orc1-5 of the yeast origin recognition complex (ORC).
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
