-検索条件
-検索結果
検索 (著者・登録者: rence & c)の結果806件中、1から50件目までを表示しています
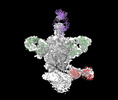
EMDB-44246: 
Cryo-EM structure of HIV-1 JRFL v6 Env in complex with vaccine-elicited, Membrane Proximal External Region (MPER) directed antibody DH1317.4.

EMDB-50321: 
sub-tomogram averaging map of CHIKV replication organelle connection to the plasma membrane
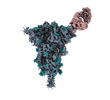
EMDB-40812: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 1

EMDB-40813: 
Structure of SARS-CoV-2 (HP-GSAS-Mut7) spike in complex with TXG-0078 Fab -Conformation 2

EMDB-44642: 
Cholecystokinin 1 receptor (CCK1R) Y140A mutant, Gq chimera (mGsqi) complex

EMDB-44643: 
Cholecystokinin 1 receptor (CCK1R) sterol 7M mutant, Gq chimera (mGsqi) complex

PDB-9bkj: 
Cholecystokinin 1 receptor (CCK1R) Y140A mutant, Gq chimera (mGsqi) complex

PDB-9bkk: 
Cholecystokinin 1 receptor (CCK1R) sterol 7M mutant, Gq chimera (mGsqi) complex

EMDB-43529: 
L5A7 Fab bound to Indonesia2005 Hemagglutinin

EMDB-43545: 
L5A7 Fab bound to 28H6E11 anti-idiotype Fab

PDB-8vue: 
L5A7 Fab bound to Indonesia2005 Hemagglutinin

PDB-8vuz: 
L5A7 Fab bound to 28H6E11 anti-idiotype Fab

EMDB-40814: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

PDB-8swh: 
Local refinement of SARS-CoV-2 (HP-GSAS-Mut7) spike NTD in complex with TXG-0078 Fab

EMDB-40815: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, N611 and base from participant 017

EMDB-40816: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 03

EMDB-40817: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp41-N611/FP and base from participant 07

EMDB-40818: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies gp120-GH and base from participant 09

EMDB-40819: 
BG505 SOSIP.664 in complex with wk26 human polyclonal antibodies C3V5, V1V3, gp41-GH/FP and base from participant 11

EMDB-43658: 
SARS-CoV-2 S (C.37 Lambda variant) plus S309, S2L20, and S2X303 Fabs

EMDB-43659: 
SARS-CoV-2 S NTD (C.37 Lambda variant) plus S2L20 and S2X303 Fabs, local refinement
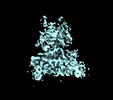
EMDB-43660: 
SARS-CoV-2 S RBD (C.37 Lambda variant) plus S309 Fab, local refinement

PDB-8vye: 
SARS-CoV-2 S (C.37 Lambda variant) plus S309, S2L20, and S2X303 Fabs

PDB-8vyf: 
SARS-CoV-2 S NTD (C.37 Lambda variant) plus S2L20 and S2X303 Fabs, local refinement

PDB-8vyg: 
SARS-CoV-2 S RBD (C.37 Lambda variant) plus S309 Fab, local refinement

EMDB-16916: 
Bipartite interaction of TOPBP1 with the GINS complex

PDB-8ok2: 
Bipartite interaction of TOPBP1 with the GINS complex

EMDB-41805: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo

PDB-8u18: 
Cryo-EM structure of murine Thrombopoietin receptor ectodomain in complex with Tpo

EMDB-43542: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation

PDB-8vuw: 
ELIC5 with cysteamine in 2:1:1 POPC:POPE:POPG nanodisc in open conformation
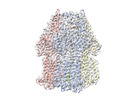
EMDB-17350: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

PDB-8p1i: 
Single particle cryo-EM co-structure of Klebsiella pneumoniae AcrB with the BDM91288 efflux pump inhibitor at 2.97 Angstrom resolution

EMDB-41510: 
Eukaryotic translation initiation factor 2B tetramer

EMDB-41566: 
Eukaryotic translation initiation factor 2B with a mutation (L516A) in the delta subunit

PDB-8tqo: 
Eukaryotic translation initiation factor 2B tetramer

PDB-8tqz: 
Eukaryotic translation initiation factor 2B with a mutation (L516A) in the delta subunit

EMDB-18004: 
Cryo electron tomogram of Caulobacter crescentus - Delta-bla

EMDB-18005: 
Cryo electron tomogram of Caulobacter crescentus - Delta-bla;tipT::Tn

EMDB-18006: 
Cryo electron tomogram of Caulobacter crescentus - Delta-bla;pSRKacrAB::nodT

EMDB-18007: 
Cryo electron tomogram of Caulobacter crescentus - Delta-bla;pSRKacrAB::nodT example 2

EMDB-18008: 
Cryo electron tomogram of Caulobacter crescentus - Delta-bla;tipR::Tn

EMDB-29452: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

PDB-8fu3: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

EMDB-17819: 
XBB 1.0 RBD bound to P4J15 (Local)
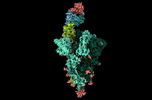
EMDB-17849: 
XBB 1.0 RBD bound to P4J15 (Global)

EMDB-17850: 
SARS-CoV-2 XBB 1.0 closed conformation.

PDB-8pq2: 
XBB 1.0 RBD bound to P4J15 (Local)

PDB-8psd: 
SARS-CoV-2 XBB 1.0 closed conformation.

EMDB-41994: 
KCTD5/Cullin3/Gbeta1gamma2 Complex: Local Refinement of KCTD5(CTD)/Gbeta1gamma2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
