-検索条件
-検索結果
検索 (著者・登録者: mori & t)の結果641件中、1から50件目までを表示しています

EMDB-36851: 
Cryo-EM structure of PseP with NAD at 2.86 angstrom resolution

PDB-8k3h: 
Cryo-EM structure of PseP with NAD at 2.86 angstrom resolution

EMDB-36852: 
Cryo-EM structure of PseP apo

PDB-8k3i: 
Cryo-EM structure of PseP apo
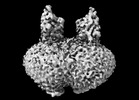
EMDB-44293: 
Cryo-EM structure of MraY in complex with analogue 2
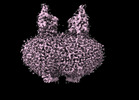
EMDB-44294: 
Cryo-EM structure of MraY in complex with analogue 3

PDB-9b70: 
Cryo-EM structure of MraY in complex with analogue 2

PDB-9b71: 
Cryo-EM structure of MraY in complex with analogue 3

EMDB-50296: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

PDB-9fbv: 
70S Escherichia coli ribosome with P-site initiatior tRNA.

EMDB-19163: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19164: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-19165: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

EMDB-19166: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rgz: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh0: 
Trimeric HSV-1F gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

PDB-8rh1: 
Trimeric HSV-2F gB ectodomain in postfusion conformation with three bound HDIT101 Fab molecules.

PDB-8rh2: 
Trimeric HSV-2G gB ectodomain in postfusion conformation with three bound HDIT102 Fab molecules.

EMDB-19851: 
Structure of the Integrator arm module containing INTS10/13/14 subunits

EMDB-19853: 
Structure of the human INTS5/8/10/15 subcomplex

EMDB-19871: 
Structure of the Integrator arm module containing INTS10/13/14/15 subunits (state 2)

EMDB-19872: 
Structure of Integrator subcomplex INTS5/8/15

EMDB-50267: 
Structure of the Integrator arm module containing subunits INTS10/13/14/15 (state 1)

EMDB-50268: 
Structure of the Integrator arm module containing subunits INTS10/13/14/15 (state 3)

PDB-9eoc: 
Structure of the Integrator arm module containing INTS10/13/14 subunits

PDB-9eof: 
Structure of the human INTS5/8/10/15 subcomplex

PDB-9ep1: 
Structure of the Integrator arm module containing INTS10/13/14/15 subunits (state 2)

PDB-9ep4: 
Structure of Integrator subcomplex INTS5/8/15

PDB-9fa4: 
Structure of the Integrator arm module containing subunits INTS10/13/14/15 (state 1)

PDB-9fa7: 
Structure of the Integrator arm module containing subunits INTS10/13/14/15 (state 3)

EMDB-38372: 
SARS-CoV-2 Omicron BQ.1.1 Variant Spike Protein Complexed with MO11 Fab

PDB-8xi6: 
SARS-CoV-2 Omicron BQ.1.1 Variant Spike Protein Complexed with MO11 Fab

EMDB-38986: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in LMNG detergent

EMDB-38987: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in nanodisc

PDB-8y6h: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in LMNG detergent

PDB-8y6i: 
P-glycoprotein in complex with UIC2 Fab and triple elacridar molecules in nanodisc

EMDB-41363: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

PDB-8tl6: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

EMDB-17171: 
CTE typeI tau filament from Guam ALS/PDC

EMDB-17173: 
CTE typeIII tau filament from Guam ALS/PDC

EMDB-17174: 
CTE typeII tau filament from Guam ALS/PDC

EMDB-17175: 
TMEM106B Fold1-s filament from Guam ALS/PDC

EMDB-17176: 
TMEM106B Fold I-d filament from Guam ALS/PDC

EMDB-17177: 
Ab typeII filament from Guam ALS/PDC

EMDB-17178: 
CTE typeI tau filament from Kii ALS/PDC

EMDB-17179: 
TypeII tau filament from Kii ALS/PDC

EMDB-17180: 
CTE typeIII tau filament

EMDB-17181: 
PHF tau filament from Kii ALS/PDC
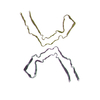
PDB-8ot6: 
CTE typeI tau filament from Guam ALS/PDC

PDB-8ot9: 
CTE typeIII tau filament from Guam ALS/PDC
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
