-検索条件
-検索結果
検索 (著者・登録者: matsuo & h)の結果134件中、1から50件目までを表示しています

EMDB-16319: 
Structure of the K+/H+ exchanger KefC with GSH

PDB-8by2: 
Structure of the K+/H+ exchanger KefC with GSH.

EMDB-16318: 
Structure of the K/H exchanger KefC

PDB-8bxg: 
Structure of the K/H exchanger KefC.

EMDB-16552: 
Structure of the RQT-bound 80S ribosome from S. cerevisiae (C2) - composite map

EMDB-16553: 
Structure of RQT (C1) bound to the stalled ribosome in a disome unit from S. cerevisiae - focused Refinement 80S

EMDB-16554: 
Structure of RQT (C1) bound to the stalled ribosome in a disome unit from S. cerevisiae - focused Refinement RQT
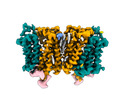
EMDB-18634: 
Cryo-EM structure of human SLC15A4 dimer in outward open state in MSP1D1 nanodisc

PDB-8qsk: 
Cryo-EM structure of human SLC15A4 dimer in outward open state in MSP1D1 nanodisc

EMDB-18637: 
Cryo-EM structure of human SLC15A4 monomer in outward open state in PMAL C8

PDB-8qsn: 
Cryo-EM structure of human SLC15A4 monomer in outward open state in PMAL C8
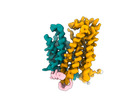
EMDB-18636: 
Cryo-EM structure of human SLC15A4 monomer in outward open state in LMNG

PDB-8qsm: 
Cryo-EM structure of human SLC15A4 monomer in outward open state in LMNG
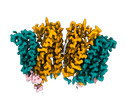
EMDB-18635: 
Cryo-EM structure of human SLC15A4 dimer in outward open state in LMNG

PDB-8qsl: 
Cryo-EM structure of human SLC15A4 dimer in outward open state in LMNG

EMDB-34412: 
Cryo-EM structure of APOBEC3G-Vif complex
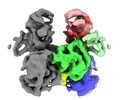
EMDB-35997: 
Cryo-EM structure of APOBEC3G-Vif complex

EMDB-35998: 
Cryo-EM structure of APOBEC3G-Vif complex

EMDB-35999: 
Cryo-EM structure of APOBEC3G-Vif complex

PDB-8h0i: 
Cryo-EM structure of APOBEC3G-Vif complex

PDB-8j62: 
Cryo-EM structure of APOBEC3G-Vif complex

EMDB-15280: 
RQT-bound 80S ribosome from S. cerevisiae (C1, raw consensus map RQT)

EMDB-15228: 
RQT-bound 80S ribosome from S. cerevisiae (C1, raw consensus map)

EMDB-16470: 
Cryo-EM structure of in vitro reconstituted Otu2-bound Ub-40S complex - body 1

EMDB-16471: 
in vitro reconstituted yeast 40S ribosome complex with deubiquitinating enzyme Otu2 bound to ubiquitinated eS7 - body 2 (40S head)

EMDB-16525: 
Cryo-EM structure of native Otu2-bound ubiquitinated 43S pre-initiation complex

EMDB-16533: 
Cryo-EM structure of native Otu2-bound ubiquitinated 48S initiation complex (partial)

EMDB-16541: 
Yeast cytoplasmic pre-40S ribosome biogenesis complex with deubiquitinating enzyme Otu2

EMDB-16542: 
Local refinement map of Otu2 N-terminal domain bound to yeast 40S ribosome

EMDB-16548: 
Local refinement map of Otu2 C-terminal domain bound to ubiquitinated eS7 on yeast 40S ribosome

PDB-8c83: 
Cryo-EM structure of in vitro reconstituted Otu2-bound Ub-40S complex

PDB-8cah: 
Cryo-EM structure of native Otu2-bound ubiquitinated 43S pre-initiation complex

PDB-8cas: 
Cryo-EM structure of native Otu2-bound ubiquitinated 48S initiation complex (partial)

PDB-8cbj: 
Cryo-EM structure of Otu2-bound cytoplasmic pre-40S ribosome biogenesis complex

EMDB-15296: 
Yeast RQC complex in state G

EMDB-15423: 
Yeast RQC complex in state F

EMDB-15424: 
Yeast RQC complex in state E

EMDB-15425: 
Yeast RQC complex in state H

EMDB-15426: 
Yeast RQC complex in state D

EMDB-15427: 
Yeast RQC complex in state with the RING domain of Ltn1 in the IN position

EMDB-15428: 
Yeast RQC complex in state with the RING domain of Ltn1 in the OUT position

EMDB-15430: 
Yeast RQC complex in state A (pre-initiation)

EMDB-15431: 
Yeast RQC complex in state B (A-site tRNA only)

EMDB-15432: 
Yeast RQC complex in state C

EMDB-15433: 
Yeast RQC complex in state I (translocation with A-site and E-site tRNAs)

EMDB-15434: 
Yeast RQC complex in state J

PDB-8aaf: 
Yeast RQC complex in state G

PDB-8agt: 
Yeast RQC complex in state F

PDB-8agu: 
Yeast RQC complex in state E

PDB-8agv: 
Yeast RQC complex in state H
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
