-検索条件
-検索結果
検索 (著者・登録者: liang & h)の結果2,388件中、1から50件目までを表示しています

EMDB-42291: 
Structure of the human INTS9-INTS11-BRAT1 complex

EMDB-42292: 
Structure of the Drosophila IntS11-CG7044(dBRAT1) complex

EMDB-60100: 
SARS-CoV-2 spike trimer (6P) in complex with three R1-26 Fabs

EMDB-60101: 
SARS-CoV-2 spike trimer (6P) in complex with R1-26 Fab, head-to-head aggregate

EMDB-60102: 
SARS-CoV-2 spike trimer (6P) in complex with R1-26 Fab, focused refinement of RBD-Fab region

EMDB-60103: 
SARS-CoV-2 spike trimer (6P) in complex with two H18 Fabs

EMDB-60104: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs

EMDB-60105: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs, head-to-head aggregate (C1 symmetry)

EMDB-60106: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs, head-to-head aggregate (C3 symmetry)

EMDB-60107: 
SARS-CoV-2 spike trimer (6P) in complex with two H18 and two R1-32 Fabs

EMDB-60108: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 and three R1-32 Fabs

EMDB-60109: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 and three R1-32 Fabs (one RBD rotated)

EMDB-60110: 
SARS-CoV-2 S1 in complex with H18 and R1-32 Fab

EMDB-60111: 
Dimer of SARS-CoV-2 S1 in complex with H18 and R1-32 Fabs
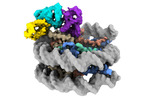
EMDB-38099: 
Cryo-EM structures of RNF168/UbcH5c-Ub in complex with H2AK13Ub nucleosomes determined by intein-based E2-Ub-NCP conjugation strategy
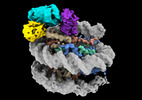
EMDB-38100: 
Cryo-EM structures of RNF168/UbcH5c-Ub/nucleosomes complex determined by activity-based chemical trapping strategy
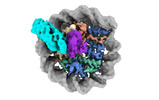
EMDB-38101: 
Cryo-EM structures of RNF168/UbcH5c-Ub in complex with H2AK13Ub nucleosomes determined by activity-based chemical trapping strategy (adjacent H2AK13/15 dual-monoubiquitination)
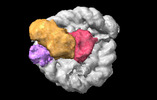
EMDB-38102: 
Cryo-EM map of RNF168/UbcH5c-Ub/nucleosome determined by E2-Ub-NCP conjugation strategy

EMDB-38873: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 50S ribosome in complex with MCX-190.

EMDB-38874: 
Cryo-EM structure of Staphylococcus aureus (15B196) 50S ribosome in complex with MCX-190.

EMDB-38875: 
Cryo-EM structure of Staphylococcus aureus 70S ribosome (strain 15B196) in complex with MCX-190.

EMDB-38876: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 70S ribosome in complex with MCX-190.

PDB-8y36: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 50S ribosome in complex with MCX-190.

PDB-8y37: 
Cryo-EM structure of Staphylococcus aureus (15B196) 50S ribosome in complex with MCX-190.

PDB-8y38: 
Cryo-EM structure of Staphylococcus aureus 70S ribosome (strain 15B196) in complex with MCX-190.

PDB-8y39: 
cryo-EM structure of Staphylococcus aureus(ATCC 29213) 70S ribosome in complex with MCX-190.

EMDB-44351: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, V1

PDB-9b8p: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, V1
EMDB-38784: 
The structure of fox ACE2 and PT RBD complex

EMDB-38792: 
The structure of fox ACE2 and SARS-CoV RBD complex

PDB-8xyz: 
The structure of fox ACE2 and PT RBD complex

PDB-8xzb: 
The structure of fox ACE2 and SARS-CoV RBD complex

EMDB-42965: 
CryoEM structure of AriA-Ocr complex

EMDB-42966: 
CryoEM structure of AriA-AriB complex (Form I)

EMDB-42967: 
CryoEM structure of AriA-AriB complex (Form II)

EMDB-42968: 
CryoEM structure of AriA-AriB complex (Form III)

EMDB-42969: 
CryoEM structure of AriA (E393Q) sensory subunit

EMDB-44350: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, Vo

EMDB-44352: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, peripheral stalks

EMDB-44353: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3

EMDB-44354: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 2

EMDB-44355: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 1

PDB-9b8o: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, Vo

PDB-9b8q: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3, peripheral stalks

PDB-9brb: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 1

PDB-9brc: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 2

PDB-9brd: 
Synaptic Vesicle V-ATPase with synaptophysin and SidK, State 3

EMDB-39858: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum bound with geranyl acetate
EMDB-39873: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum

PDB-8z9a: 
Cryo-EM structure of the insect olfactory receptor OR5-Orco heterocomplex from Acyrthosiphon pisum bound with geranyl acetate
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
