-検索条件
-検索結果
検索 (著者・登録者: he & x)の結果16,171件中、1から50件目までを表示しています

EMDB-44372: 
In-cell Saccharomyces cerevisiae nuclear pore complex with single nuclear ring

EMDB-44377: 
In-cell Saccharomyces cerevisiae nuclear pore complex with double nuclear ring and basket

EMDB-44379: 
In-cell Mus musculus nuclear pore complex with nuclear basket

EMDB-44381: 
In-cell Toxoplasma gondii nuclear pore complex

EMDB-45197: 
In-cell Saccharomyces cerevisiae symmetry-expanded nuclear pore complex with double nuclear ring and basket

EMDB-45198: 
In-cell Saccharomyces cerevisiae symmetry-expanded nuclear pore complex with single nuclear ring

EMDB-45199: 
In-cell Saccharomyces cerevisiae nuclear pore complex cytoplasmic ring focused refinement

EMDB-45200: 
In-cell Saccharomyces cerevisiae nuclear pore complex inner ring focused refinement

EMDB-45201: 
In-cell Saccharomyces cerevisiae nuclear pore complex single nuclear ring focused refinement

EMDB-45202: 
In-cell Saccharomyces cerevisiae nuclear pore complex double nuclear ring focused refinement

EMDB-45203: 
In-cell Saccharomyces cerevisiae nuclear pore complex nuclear basket focused refinement

EMDB-45204: 
In-cell Saccharomyces cerevisiae nuclear pore complex membrane focused refinement for single nuclear ring

EMDB-45205: 
In-cell Saccharomyces cerevisiae nuclear pore complex membrane focused refinement for double nuclear ring

EMDB-45216: 
In-cell Mus musculus nuclear pore complex with nuclear basket consensus map

EMDB-45219: 
In-cell Mus musculus nuclear pore complex cytoplasmic ring focused refinement

EMDB-45220: 
In-cell Mus musculus nuclear pore complex inner ring focused refinement

EMDB-45222: 
In-cell Mus musculus nuclear pore complex nuclear ring focused refinement

EMDB-45223: 
In-cell Mus musculus nuclear pore complex basket focused refinement

EMDB-45227: 
In-cell Mus musculus nuclear pore complex membrane focused refinement

EMDB-45228: 
In-cell Toxoplasma gondii symmetry-expanded nuclear pore complex
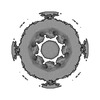
EMDB-45255: 
In-cell Saccharomyces cerevisiae C8-symmetrised nuclear pore complex consensus map

EMDB-45256: 
In-cell Saccharomyces cerevisiae symmetry-expanded nuclear pore complex consensus map

EMDB-45257: 
In-cell Mus musculus nuclear pore complex with nuclear basket consensus map

EMDB-45258: 
In-cell Mus musculus symmetry-expanded nuclear pore complex with nuclear basket consensus map

EMDB-45259: 
In-cell Toxoplasma gondii C8-symmetrised nuclear pore complex consensus map

EMDB-41766: 
CryoEM structure of D2 dopamine receptor in complex with GoA KE mutant, scFv16, and dopamine

EMDB-41776: 
CryoEM structure of D2 dopamine receptor in complex with GoA KE mutant and dopamine

EMDB-44551: 
Map of eastern equine encephalitis virus q3 spike protein in complex with VLDLR without masked refinement

EMDB-45206: 
Reconstituted P400 Subcomplex of the human TIP60 complex

EMDB-45240: 
P400 subcomplex of the native human TIP60 complex

EMDB-45252: 
ARP module of the human TIP60 complex
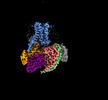
EMDB-60628: 
Carazolol-activated human beta3 adrenergic receptor
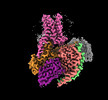
EMDB-60629: 
Epinephrine-activated human beta3 adrenergic receptor

EMDB-45962: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 5-12-18

EMDB-45963: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18

EMDB-45964: 
Cryo-EM structure of Tulane virus 9-6-17 variant capsid protein VP1 9-14-18, DTT-treated

EMDB-31367: 
Structure of mumps virus nucleoprotein without C-arm

EMDB-44486: 
Structure of apo-state V. cholerae DdmD

EMDB-45254: 
Structure of V. cholerae monomeric DdmD bound with ssDNA

EMDB-60410: 
Cryo-EM structure of human ZnT1, in the absence of zinc, determined in outward-facing conformation

EMDB-60435: 
Cryo-EM structure of human ZnT1, in the presence of zinc, determined in outward-facing conformation

EMDB-42284: 
The structure of the Clostridium thermocellum AdhE spirosome

EMDB-60099: 
SARS-CoV-2 spike trimer (6P) in complex with two R1-26 Fabs

EMDB-60100: 
SARS-CoV-2 spike trimer (6P) in complex with three R1-26 Fabs

EMDB-60101: 
SARS-CoV-2 spike trimer (6P) in complex with R1-26 Fab, head-to-head aggregate

EMDB-60102: 
SARS-CoV-2 spike trimer (6P) in complex with R1-26 Fab, focused refinement of RBD-Fab region

EMDB-60103: 
SARS-CoV-2 spike trimer (6P) in complex with two H18 Fabs

EMDB-60104: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs

EMDB-60105: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs, head-to-head aggregate (C1 symmetry)

EMDB-60106: 
SARS-CoV-2 spike trimer (6P) in complex with three H18 Fabs, head-to-head aggregate (C3 symmetry)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
