-検索条件
-検索結果
検索 (著者・登録者: hale & c)の結果235件中、1から50件目までを表示しています

EMDB-18438: 
mt-SSU assembly intermediate in GTPBP8 knock-out cells, state 1

EMDB-18439: 
mt-SSU assembly intermediate in GTPBP8 knock-out cells, state 2

EMDB-18440: 
mt-SSU assembly intermediate in GTPBP8 knock-out cells, state 3

EMDB-18443: 
mt-SSU assembly intermediate in GTPBP8 knock-out cells, state 4

EMDB-18460: 
mt-LSU assembly intermediate in GTPBP8 knock-out cells, state 1

EMDB-18461: 
mt-LSU assembly intermediate in GTPBP8 knock-out cells, state 2

EMDB-43275: 
GluA2 bound to GYKI-52466 and Glutamate, Inhibited State 1

EMDB-43276: 
GluA2 bound to GYKI-52466 and Glutamate, Inhibited State 2

PDB-8vj6: 
GluA2 bound to GYKI-52466 and Glutamate, Inhibited State 1

PDB-8vj7: 
GluA2 bound to GYKI-52466 and Glutamate, Inhibited State 2

EMDB-18826: 
In situ sub-tomogram average of the E. coli 70S ribosome obtained using honeycomb gold supports

EMDB-43931: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

EMDB-43932: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex

PDB-9axa: 
CryoEM structure of activated CRAF/MEK/14-3-3 complex with NST-628

PDB-9axc: 
Activated CRAF/MEK heterotetramer from focused refinement of CRAF/MEK/14-3-3 complex

EMDB-15769: 
A gap across the beta rings in 20S proteasome

EMDB-15767: 
Bovine 20S proteasome, untreated

EMDB-15768: 
Partially disassembled 20S proteasome upon disulfide bond formation.

PDB-8azk: 
Bovine 20S proteasome, untreated

EMDB-42601: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1

EMDB-42480: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

EMDB-42484: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain

PDB-8ur3: 
Cryo-EM reconstruction of Staphylococcus aureus Oleate hydratase (OhyA) dimer with an ordered C-terminal membrane-association domain

PDB-8ur6: 
Cryo-EM reconstruction of Staphylococcus aureus oleate hydratase (OhyA) dimer with a disordered C-terminal membrane-association domain

EMDB-29026: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1

PDB-8feg: 
CryoEM structure of Kappa Opioid Receptor bound to a semi-peptide and Gi1
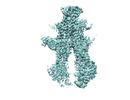
EMDB-41004: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP2

PDB-8t3k: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP2
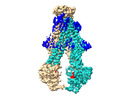
EMDB-29362: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP

PDB-8fpf: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to ATP: BmrCD_IF-ATP

EMDB-29087: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ATP: BmrCD_OC-ATP

EMDB-29297: 
Structure0915

EMDB-40908: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-1HT/ATP

EMDB-40974: 
BmrCD_OC-ADPVi

EMDB-41058: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ADPVi: BmrCD_IF-HT/ADPVi

PDB-8fhk: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ATP: BmrCD_OC-ATP

PDB-8fmv: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-2HT/ATP

PDB-8szc: 
Heterodimeric ABC transporter BmrCD in the inward-facing conformation bound to substrate and ATP: BmrCD_IF-1HT/ATP

PDB-8t1p: 
Heterodimeric ABC transporter BmrCD in the occluded conformation bound to ADPVi: BmrCD_OC-ADPVi

EMDB-17756: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

PDB-8pm2: 
Structure of the murine trace amine-associated receptor TAAR7f bound to N,N-dimethylcyclohexylamine (DMCH) in complex with mini-Gs trimeric G protein

EMDB-15786: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli (Apo form)

EMDB-15787: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE

EMDB-15788: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE (C387S mutant)

EMDB-15789: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Lyso-PE

EMDB-15790: 
Cryo-EM structure apolipoprotein N-acyltransferase Lnt from E.coli in complex with FP3

EMDB-15791: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Pam3

PDB-8b0k: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli (Apo form)

PDB-8b0l: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE

PDB-8b0m: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE (C387S mutant)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
