-検索条件
-検索結果
検索 (著者・登録者: grob & p)の結果78件中、1から50件目までを表示しています

EMDB-43893: 
Structure of the auto-fluorescent membrane-bound red body organelle from Nannochloropsis oceanica in situ

EMDB-40554: 
Cryo-EM Consensus map of the E. coli transcription-translation complex (RNAP in an anti-swiveled conformation)

EMDB-40178: 
Cryo-EM composited map of the E. coli transcription-translation complex (RNAP in an anti-swiveled conformation)
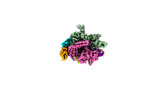
EMDB-29212: 
Cryo-EM structure of E. coli RNA polymerase backtracked elongation complex harboring a terminal mismatch

EMDB-29213: 
Cryo-EM structure of E. coli RNA polymerase Elongation complex in the Transcription-Translation Complex (RNAP in an anti-swiveled conformation)
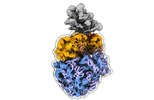
EMDB-29214: 
Cryo-EM structure of E. coli 70S Ribosome containing mRNA and tRNA (in the transcription-translation complex)

PDB-8fix: 
Cryo-EM structure of E. coli RNA polymerase backtracked elongation complex harboring a terminal mismatch

PDB-8fiy: 
Cryo-EM structure of E. coli RNA polymerase Elongation complex in the Transcription-Translation Complex (RNAP in an anti-swiveled conformation)

PDB-8fiz: 
Cryo-EM structure of E. coli 70S Ribosome containing mRNA and tRNA (in the transcription-translation complex)

EMDB-27112: 
S728-1157 IgG in complex with SARS-CoV-2-6P-Mut7 Spike protein (global refinement)

EMDB-27113: 
S728-1157 IgG in complex with SARS-CoV-2-6P-Mut7 Spike protein (focused refinement)

PDB-8d0z: 
S728-1157 IgG in complex with SARS-CoV-2-6P-Mut7 Spike protein (focused refinement)

EMDB-25107: 
Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle by localized reconstruction

EMDB-25108: 
I53-50 nanoparticle core reconstructed from GPC-I53-50NP by focused refinement

EMDB-25109: 
Lassa virus glycoprotein construct (Josiah GPC-I53-50A) in complex with LAVA01 antibody

PDB-7sgd: 
Lassa virus glycoprotein construct(Josiah GPCysR4) recovered from GPC-I53-50 nanoparticle by localized reconstruction

PDB-7sge: 
I53-50 nanoparticle core reconstructed from GPC-I53-50NP by focused refinement

PDB-7sgf: 
Lassa virus glycoprotein construct (Josiah GPC-I53-50A) in complex with LAVA01 antibody
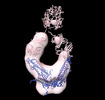
EMDB-26217: 
Negative stain EM map of COVA1-07 mAb bound to the S2 domain of SARS-CoV-2 S
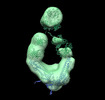
EMDB-26218: 
Negative stain EM map of COVA2-14 mAb bound to the S2 domain of SARS-CoV-2 S

EMDB-26219: 
Negative stain EM map of COVA2-18 mAb bound to the S2 domain of SARS-CoV-2 S
EMDB-26220: 
Negative stain EM map of the S2 domain of SARS-CoV-2 S

EMDB-13776: 
Structure of formaldehyde cross-linked SARS-CoV-2 S glycoprotein

PDB-7q1z: 
Structure of formaldehyde cross-linked SARS-CoV-2 S glycoprotein

EMDB-13185: 
Helical structure of the toxin MakA from Vibrio cholera

EMDB-25634: 
Negative stain map of monoclonal Fab 047-09 4F04 binding the anchor epitope of H1 HA

EMDB-25635: 
Negative stain map of monoclonal Fab 241 IgA 2F04 binding the anchor epitope of H1 HA

EMDB-25636: 
Negative stain map of polyclonal Fab 236.7 binding the anchor and esterase epitopes of H1 HA

EMDB-25637: 
Negative stain map of polyclonal Fab 236.7 binding the RBS epitope of H1 HA

EMDB-25638: 
Negative stain map of polyclonal Fab 236.14 binding an epitope on the top of the head of H1 HA
EMDB-25639: 
Negative stain map of polyclonal Fab 236.14 binding the esterase epitope of H1 HA

EMDB-25640: 
Negative stain map of polycolonal Fab 236.14 binding the RBS epitope of H1 HA

EMDB-25641: 
Negative stain map of polyclonal Fab 236.14 binding the anchor epitope of H1 HA
EMDB-25642: 
Negative stain map of polyclonal Fab 241.7 binding the esterase epitope of H1 HA
EMDB-25643: 
Negative stain map of polyclonal Fab 241.14 binding the anchor epitope of H1 HA
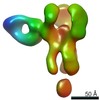
EMDB-25644: 
Negative stain map of polyclonal Fab 241.14 binding the esterase epitope of H1 HA

EMDB-25645: 
Negative stain map of polyclonal Fab 241.14 binding an epitope on the top of the head of H1 HA

EMDB-25646: 
Negative stain map of polyclonal Fab 241.14 binding the RBS epitope of H1 HA

EMDB-25655: 
CryoEM map of anchor 222-1C06 Fab and lateral patch 2B05 Fab binding H1 HA

PDB-7t3d: 
CryoEM map of anchor 222-1C06 Fab and lateral patch 2B05 Fab binding H1 HA

EMDB-23792: 
CryoEM structure of monoclonal Fab 045-09 2B05 binding the lateral patch of influenza virus H1 HA
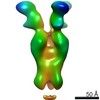
EMDB-23793: 
Negative stain map of monoclonal Fab SFV009 2G01 binding the RBS of H1 HA

EMDB-23794: 
Negative stain map of monoclonal Fab 045-09 2B05 binding the lateral patch of H1 HA

EMDB-23795: 
Negative stain map of monoclonal Fab SFV019 2A06 binding the lateral patch of H1 HA
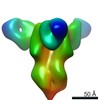
EMDB-23796: 
Negative stain map of monoclonal Fab SFV015 2F02 binding the lateral patch of H1 HA

EMDB-23797: 
Negative stain map of monoclonal Fab 047-09 4G02 binding the lateral patch of H1 HA
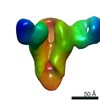
EMDB-23798: 
Negative stain map of monoclonal Fab 047-09 4B06 binding the lateral patch of H1 HA

PDB-7mem: 
CryoEM structure of monoclonal Fab 045-09 2B05 binding the lateral patch of influenza virus H1 HA

EMDB-9107: 
A unique supramolecular organization of photosystem I in the moss Physcomitrella patens

PDB-6mem: 
A unique supramolecular organization of photosystem I in the moss Physcomitrella patens
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
