-検索条件
-検索結果
検索 (著者・登録者: georgescu & r)の結果71件中、1から50件目までを表示しています

EMDB-41252: 
Cryo-EM map of the Saccharomyces cerevisiae PCNA clamp unloader Elg1-RFC complex

EMDB-41253: 
Cryo-EM map of the Saccharomyces cerevisiae clamp unloader Elg1-RFC bound to a cracked PCNA

EMDB-41254: 
Cryo-EM map of the Saccharomyces cerevisiae clamp unloader Elg1-RFC bound to PCNA

PDB-8thb: 
Structure of the Saccharomyces cerevisiae PCNA clamp unloader Elg1-RFC complex

PDB-8thc: 
Structure of the Saccharomyces cerevisiae clamp unloader Elg1-RFC bound to a cracked PCNA

PDB-8thd: 
Structure of the Saccharomyces cerevisiae clamp unloader Elg1-RFC bound to PCNA

EMDB-29412: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 1 (open 9-1-1 and shoulder bound DNA only)

EMDB-29413: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 2 (open 9-1-1 ring and flexibly bound chamber DNA)

EMDB-29414: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 3 (open 9-1-1 and stably bound chamber DNA)

EMDB-29415: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 4 (partially closed 9-1-1 and stably bound chamber DNA)

EMDB-29416: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 5 (closed 9-1-1 and stably bound chamber DNA)

EMDB-29417: 
EM map of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 5-nt gapped DNA (9-1-1 encircling fully bound DNA)

PDB-8fs3: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 1 (open 9-1-1 and shoulder bound DNA only)

PDB-8fs4: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 2 (open 9-1-1 ring and flexibly bound chamber DNA)

PDB-8fs5: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 3 (open 9-1-1 and stably bound chamber DNA)

PDB-8fs6: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 4 (partially closed 9-1-1 and stably bound chamber DNA)

PDB-8fs7: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 10-nt gapped DNA in step 5 (closed 9-1-1 and stably bound chamber DNA)

PDB-8fs8: 
Structure of S. cerevisiae Rad24-RFC loading the 9-1-1 clamp onto a 5-nt gapped DNA (9-1-1 encircling fully bound DNA)
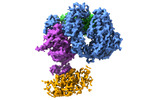
EMDB-29345: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase in Apo state conformation I
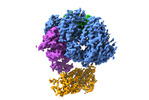
EMDB-29346: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in Apo state conformation II

EMDB-29347: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex bound to a template DNA
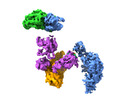
EMDB-29349: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the RNA synthesis state
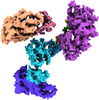
EMDB-29351: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the post RNA handoff state
EMDB-29352: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the DNA elongation state

PDB-8foc: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase in Apo state conformation I

PDB-8fod: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in Apo state conformation II

PDB-8foe: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex bound to a template DNA

PDB-8foh: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the RNA synthesis state

PDB-8foj: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the post RNA handoff state

PDB-8fok: 
Cryo-EM structure of S. cerevisiae DNA polymerase alpha-primase complex in the DNA elongation state

EMDB-28195: 
SV40 T-Antigen helicase hexamer

EMDB-25872: 
Cryo-EM 3D map of the S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to two DNA molecules, one at the 5'-recessed end and the other at the 3'-recessed end

EMDB-25873: 
Cryo-EM 3D map of the S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to DNA with an open clamp

EMDB-25874: 
Cryo-EM 3D map of S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to DNA with a closed clamp ring

EMDB-25875: 
Cryo-EM 3D map of S. cerevisiae clamp loader RFC bound to two DNA molecules, one at the 5'-recessed end and the other at the 3'-recessed end

EMDB-25876: 
Cryo-EM 3D map of S. cerevisiae clamp loader RFC bound to DNA

PDB-7tfh: 
Atomic model of the S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to two DNA molecules, one at the 5'-recessed end and the other at the 3'-recessed end

PDB-7tfi: 
Atomic model of the S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to DNA with an open clamp

PDB-7tfj: 
Atomic model of S. cerevisiae clamp-clamp loader complex PCNA-RFC bound to DNA with a closed clamp ring

PDB-7tfk: 
Atomic model of S. cerevisiae clamp loader RFC bound to two DNA molecules, one at the 5'-recessed end and the other at the 3'-recessed end

PDB-7tfl: 
Atomic model of S. cerevisiae clamp loader RFC bound to DNA

EMDB-25121: 
Cryo-EM 3D map of the yeast Rad24-RFC loader bound to DNA and the closed 9-1-1 clamp

EMDB-25122: 
Cryo-EM 3D map of the yeast Rad24-RFC loader bound to DNA and the open 9-1-1 clamp

PDB-7sgz: 
Structure of the yeast Rad24-RFC loader bound to DNA and the closed 9-1-1 clamp

PDB-7sh2: 
Structure of the yeast Rad24-RFC loader bound to DNA and the open 9-1-1 clamp

EMDB-22803: 
Cryo-EM 3D map of the Saccharomyces cerevisiae replicative polymerase delta in complex with a primer/template and the PCNA clamp

PDB-7kc0: 
Structure of the Saccharomyces cerevisiae replicative polymerase delta in complex with a primer/template and the PCNA clamp

EMDB-21701: 
Structure of the Saccharomyces cerevisiae polymerase epsilon holoenzyme

PDB-6wjv: 
Structure of the Saccharomyces cerevisiae polymerase epsilon holoenzyme

EMDB-20607: 
Structure of the S. cerevisiae replicative helicase CMG in complex with a forked DNA
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
