-検索条件
-検索結果
検索 (著者・登録者: davis & jh)の結果56件中、1から50件目までを表示しています

EMDB-40825: 
10E8-GT10.2 immunogen in complex with human Fab 10E8 and mouse Fab W6-10
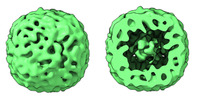
EMDB-43285: 
Human heavy-chain holoferritin refined in C1 from cryo-ET data filtered by tomoDRGN

EMDB-43286: 
Human heavy-chain apoferritin refined in C1 from cryo-ET data filtered by tomoDRGN

EMDB-43287: 
Membrane-associated 70S ribosome in complex with SecDF

EMDB-27941: 
Cryo-EM structure of substrate-free DNClpX.ClpP from singly capped particles
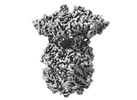
EMDB-27946: 
Cryo-EM structure of substrate-free DNClpX.ClpP

EMDB-27952: 
Cryo-EM structure of substrate-free ClpX.ClpP

EMDB-28692: 
30S_delta_ksgA_h44_inactive_conformation
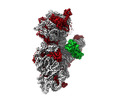
EMDB-28720: 
30S_delta_ksgA+KsgA complex

EMDB-28585: 
Cryo-EM structure of a delivery complex containing the SspB adaptor, an ssrA-tagged substrate, and the AAA+ ClpXP protease

EMDB-26792: 
Transcription co-activator SAGA

EMDB-26803: 
Cryo-EM structure of the SAGA Tra1 module

EMDB-26804: 
Cryo-EM structure of the SAGA core module

EMDB-14153: 
SARS-CoV-2 Spike, C3 symmetry

EMDB-14152: 
SARS-CoV-2 Spike with ethylbenzamide-tri-iodo Siallyllactose, C3 symmetry

EMDB-14154: 
SARS-CoV-2 Spike with ethylbenzamide-tri-iodo Siallyllactose, C1 symmetry

EMDB-14155: 
SARS-CoV-2 Spike, C1 symmetry

EMDB-25471: 
Structure of EBOV GP lacking the mucin-like domain with 1C11 scFv and 1C3 Fab bound

EMDB-22475: 
Composite cryo-EM density map of the radial spoke 1 isolated from Chlamydomonas reinhardtii

EMDB-22480: 
Cryo-EM density map of stalk of radial spoke 1 attached with doublet microtubule from Chlamydomonas reinhardtii

EMDB-22481: 
Composite cryo-EM density map of radial spoke 2 stalk, IDAc, and N-DRC attached with doublet microtubule

EMDB-22482: 
Cryo-EM density map of on-doublet radial spoke 1 head

EMDB-22483: 
Cryo-EM density map of on-doublet radial spoke 2 head

EMDB-22486: 
Cryo-EM density map of RSP1 dimer from two radial spokes

EMDB-0481: 
Role of Era in Assembly and Homeostasis of the Ribosomal Small Subunit

EMDB-0482: 
Role of Era in Assembly and Homeostasis of the Ribosomal Small Subunit

EMDB-0483: 
Role of Era in Assembly and Homeostasis of the Ribosomal Small Subunit

EMDB-0484: 
Role of Era in Assembly and Homeostasis of the Ribosomal Small Subunit

EMDB-8731: 
Influenza hemagglutinin (HA) trimer reconstruction at 4.2 Angstrom resolution using particles from micrographs tilted at 40 degrees

EMDB-8434: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class A

EMDB-8440: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class B

EMDB-8441: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class C

EMDB-8442: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class C1

EMDB-8443: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class C2

EMDB-8444: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class C3

EMDB-8445: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class D

EMDB-8446: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class D1

EMDB-8447: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class D2

EMDB-8448: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class D3

EMDB-8449: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class D4

EMDB-8450: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E

EMDB-8451: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E1

EMDB-8452: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E2

EMDB-8453: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E3

EMDB-8455: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E4

EMDB-8456: 
Structure of bL17-depleted large ribosomal subunit assembly intermediate - Class E5

EMDB-8457: 
Structure of 30S Ribosome - Class F

EMDB-8274: 
YphC and YsxC GTPases assist the maturation of the central protuberance, GTPase-associated region, and functional core of the 50S ribosomal subunit

EMDB-8275: 
YphC and YsxC GTPases assist the maturation of the central protuberance, GTPase-associated region, and functional core of the 50S ribosomal subunit

EMDB-8276: 
YphC and YsxC GTPases assist the maturation of the central protuberance, GTPase-associated region, and functional core of the 50S ribosomal subunit
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
