-検索条件
-検索結果
検索 (著者・登録者: dai & xh)の結果全48件を表示しています

EMDB-27045: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis of Neutralization Efficacy

EMDB-27047: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis of Neutralization Efficacy

EMDB-27048: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis of Neutralization Efficacy

EMDB-27052: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27049: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27050: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27051: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27053: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27054: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27055: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-27056: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy
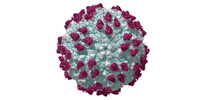
EMDB-27057: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy
EMDB-27058: 
Structures of Zika Virus in Complex with Antibodies Targeting E Dimer Epitopes and Basis for Neutralization Efficacy

EMDB-34585: 
Molecular recognition of two endogenous hormones by the human parathyroid hormone receptor-1

EMDB-34587: 
PTHrP-PTH1R-Gs complex

EMDB-34598: 
Human parathyroid hormone receptor-1 dimer

EMDB-24642: 
SARS-CoV-2 Spike bound to Fab PDI 210

EMDB-24643: 
SARS-CoV-2 Spike bound to Fab PDI 96

EMDB-24644: 
SARS-CoV-2 Spike bound to Fab PDI 215

EMDB-24645: 
SARS-CoV-2 Spike bound to Fab WCSL 119

EMDB-24646: 
SARS-CoV-2 Spike bound to Fab WCSL 129

EMDB-24647: 
SARS-CoV-2 Spike bound to Fab PDI 93

EMDB-24648: 
SARS-CoV-2 Spike bound to Fab PDI 222

EMDB-24649: 
SARS-CoV-2 receptor binding domain bound to Fab PDI 222

PDB-7rr0: 
SARS-CoV-2 receptor binding domain bound to Fab PDI 222

EMDB-21505: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21506: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21507: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21508: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21510: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21515: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-21527: 
Structures of Capsid and Capsid-Associated Tegument Complex inside the Epstein-Barr Virus

EMDB-9366: 
Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development

EMDB-9367: 
Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development

EMDB-9368: 
Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development

EMDB-9369: 
Atomic structures and deletion mutant reveal different capsid-binding patterns and functional significance of tegument protein pp150 in murine and human cytomegaloviruses with implications for therapeutic development

EMDB-7472: 
CryoEM structure of herpes simplex virus 1 capsid with associated tegument protein complexes.

EMDB-7473: 
Subparticle refinement of HSV-1 capsid vertex region.

EMDB-7047: 
CryoEM structure and atomic model of the Kaposi's sarcoma-associated herpesvirus capsid

EMDB-7034: 
Improved cryoEM structure of human adenovirus type 5 with atomic details of minor proteins VI and VII

EMDB-8397: 
In situ structures of the genome and genome-delivery apparatus in ssRNA bacteriophage MS2

EMDB-6212: 
CryoEM reconstruction of Kaposi's sarcoma-associated herpesvirus SCP-null mutant (KSHV-SCPnull)

EMDB-6213: 
CryoEM reconstruction of Kaposi's sarcoma-associated herpesvirus mutant with C-terminal half of SCP truncated (KSHV-SCPN86)

EMDB-6214: 
CryoEM reconstruction of Kaposi's sarcoma-associated herpesvirus mutant with N-terminal 6 residues of SCP truncated (KSHV-SCPdelN6)

EMDB-6038: 
Electron cryo-microscopy of Kaposi's sarcoma-associated herpesvirus virion

EMDB-5695: 
Electron cryo-microscopy of human cytomegalovirus capsid

EMDB-5696: 
Electron cryo-microscopy of human cytomegalovirus virion

EMDB-5697: 
Electron cryo-microscopy of human cytomegalovirus SCP-deficient particles
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
