-検索条件
-検索結果
検索 (著者・登録者: cho & hj)の結果55件中、1から50件目までを表示しています

EMDB-35010: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein

EMDB-35770: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Consensus map)

EMDB-35772: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (Receptor-focused map)

EMDB-35773: 
Human Consensus Olfactory Receptor OR52c in Complex with Octanoic Acid (OCA) and G Protein (G protein-focused map)
EMDB-35971: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c-bRIL
EMDB-37336: 
Human Consensus Olfactory Receptor OR52c in apo state, OR52c only

EMDB-33274: 
Human Cx36/GJD2 (N-terminal deletion mutant) gap junction channel in soybean lipids (D6 symmetry)

EMDB-33275: 
Human Cx36/GJD2 (N-terminal deletion mutant) gap junction channel in soybean lipids (C1 symmetry)

EMDB-33734: 
Cryo-EM structure of SARS-CoV-2 spike in complex with K202.B bispecific antibody

EMDB-27943: 
9H2 Fab-poliovirus 1 complex
EMDB-27947: 
9H2 Fab-Sabin poliovirus 3 complex

EMDB-27948: 
9H2 Fab-poliovirus 2 complex
EMDB-27949: 
9H2 Fab-Sabin poliovirus 3 complex
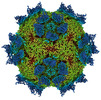
EMDB-27950: 
9H2 Fab-Sabin poliovirus 2 complex
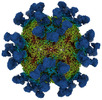
EMDB-27951: 
9H2 Fab-Sabin poliovirus 1 complex

EMDB-40455: 
Cryo-EM structure of Karyopherin-beta2 bound to HNRNPH2 PY-NLS

EMDB-33326: 
Human Cx36/GJD2 gap junction channel with pore-lining N-terminal helices in the presence of Magnesium

EMDB-33256: 
Human Cx36/GJD2 gap junction channel in detergents

EMDB-33254: 
Human Cx36/GJD2 (N-terminal deletion BRIL-fused mutant) gap junction channel in soybean lipids (D6 symmetry)

EMDB-33255: 
Human Cx36/GJD2 (N-terminal deletion BRIL-fused mutant) gap junction channel in soybean lipids (C1 symmetry)

EMDB-33270: 
Human Cx36/GJD2 (BRIL-fused mutant) gap junction channel in detergents at 2.2 Angstroms resolution

EMDB-33315: 
Human Cx36/GJD2 gap junction channel with pore-lining N-terminal helices in soybean lipids

EMDB-33327: 
Structurally hetero-junctional human Cx36/GJD2 gap junction channel in soybean lipids (C6 symmetry)

EMDB-33328: 
Structurally hetero-junctional human Cx36/GJD2 gap junction channel in soybean lipids (C1 symmetry)

EMDB-34822: 
Human Cx36/GJD2 (BRIL-fused mutant) gap junction channel in soybean lipids

EMDB-34856: 
Structurally hetero-junctional human Cx36/GJD2 gap junction channel in detergents (C6 symmetry)

EMDB-34857: 
Structurally hetero-junctional human Cx36/GJD2 gap junction channel in detergents (C1 symmetry)
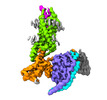
EMDB-33984: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi

EMDB-33985: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi

EMDB-35494: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Consensus map)

EMDB-35495: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Focused map on NPY-Y2R)

EMDB-35496: 
Complex structure of Neuropeptide Y Y2 receptor in complex with NPY and Gi (Focused map on Gi-scFv16)

EMDB-35497: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi (Consensus map)

EMDB-35498: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi (Focused map on PYY(3-36)-Y2R)

EMDB-35499: 
Complex structure of Neuropeptide Y Y2 receptor in complex with PYY(3-36) and Gi (Focused map on Gi-scFv16)

EMDB-25699: 
VFLIP Spike Trimer with GAR03

EMDB-25700: 
VFLIP Spike Trimer with GAR05 FAB

EMDB-26627: 
Human TMEM175 in an closed state

EMDB-26626: 
Human TMEM175 in an open state

EMDB-25408: 
Mfd DNA complex

EMDB-12775: 
Cryo-EM structure of the plectasin fibril (double strands)

EMDB-12776: 
Cryo-EM structure of the plectasin fibril (single strand)

EMDB-26216: 
Methylococcus capsulatus methane monooxygenase hydroxylase (MMOH) using the plasma-jet treated grid

EMDB-31979: 
Neuropeptide Y Y1 Receptor (NPY1R) in Complex with G Protein and its endogeneous Peptide-Agonist Neuropeptide Y (NPY)

EMDB-23775: 
Structure of human p97 ATPase L464P mutant

EMDB-23776: 
Structure of human p97 ATPase L464P mutant

EMDB-22989: 
Structure of kinase and Central lobes of yeast CKM

EMDB-22990: 
Structure of the H-lobe of yeast CKM

EMDB-22991: 
Structure of the yeast CKM

EMDB-22221: 
SARS-CoV-2 HexaPro S One RBD up
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
