-検索条件
-検索結果
検索 (著者・登録者: chait & bt)の結果69件中、1から50件目までを表示しています

EMDB-40699: 
SARS-CoV-2 replication-transcription complex bound to nsp9 and UMPCPP, as a pre-catalytic NMPylation intermediate

EMDB-40707: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9, as a noncatalytic RNA-nsp9 binding mode

EMDB-40708: 
SARS-CoV-2 replication-transcription complex bound to RNA-nsp9 and GDP-betaS, as a pre-catalytic deRNAylation/mRNA capping intermediate
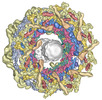
EMDB-41123: 
Composite multiscale structure of the yeast NPC

EMDB-41114: 
Nuclear double outer ring of the yeast NPC

EMDB-41116: 
Lumenal ring of the isolated yeast NPC

EMDB-41117: 
Pom34-Pom152 membrane attachment site yeast NPC

EMDB-41119: 
Cytoplasmic outer ring of the yeast NPC

EMDB-41120: 
Central transporter/plug yeast NPC

EMDB-41121: 
Double outer ring connectors of the yeast NPC

EMDB-41122: 
Connectors in cytoplasmic outer ring of yeast NPC

EMDB-41285: 
Double nuclear outer ring of Nup84-complexes from the yeast NPC

EMDB-41300: 
Inner spoke ring of the yeast NPC

EMDB-26639: 
SARS-CoV-2 replication-transcription complex bound to Remdesivir triphosphate, in a pre-catalytic state

EMDB-26641: 
SARS-CoV-2 replication-transcription complex bound to ATP, in a pre-catalytic state

EMDB-26642: 
SARS-CoV-2 replication-transcription complex bound to UTP, in a pre-catalytic state.

EMDB-26645: 
SARS-CoV-2 replication-transcription complex bound to GTP, in a pre-catalytic state

EMDB-26646: 
SARS-CoV-2 replication-transcription complex bound to CTP, in a pre-catalytic state
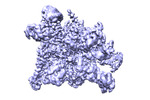
EMDB-33466: 
cryo-EM structure of HK022 putRNA-associated E.coli RNA polymerase elongation complex

EMDB-33468: 
cryo-EM structure of HK022 putRNA-less E.coli RNA polymerase elongation complex
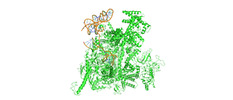
EMDB-33470: 
Cryo-EM structure of sigma70 bound HK022 putRNA-associated E.coli RNA polymerase elongation complex

EMDB-24224: 
Combined 3D structure of the isolated yeast NPC

EMDB-24225: 
Low resolution, multi-part 3D structure of the isolated yeast NPC

EMDB-24231: 
Double nuclear outer ring from the isolated yeast NPC

EMDB-24232: 
Inner ring spoke from the isolated yeast NPC

EMDB-24258: 
Structure of the in situ yeast NPC

EMDB-24107: 
Cryo-EM structure of TACAN in the apo form (TMEM120A)

EMDB-24108: 
Cryo-EM structure of TACAN in the H196A H197A mutant form (TMEM120A)

EMDB-23007: 
Structure of SARS-CoV-2 backtracked complex bound to nsp13 helicase - nsp13(1)-BTC

EMDB-23008: 
Structure of SARS-CoV-2 backtracked complex complex bound to nsp13 helicase - nsp13(2)-BTC

EMDB-23009: 
Structure of SARS-CoV-2 backtracked complex complex bound to nsp13 helicase - BTC (local refinement)

EMDB-21996: 
Mfd-bound E.coli RNA polymerase elongation complex - L1 state

EMDB-22006: 
Mfd-bound E.coli RNA polymerase elongation complex - L2 state

EMDB-22012: 
Mfd-bound E.coli RNA polymerase elongation complex - I state

EMDB-22039: 
Mfd-bound E.coli RNA polymerase elongation complex - II state

EMDB-22043: 
Mfd-bound E.coli RNA polymerase elongation complex - III state

EMDB-22044: 
Mfd-bound E.coli RNA polymerase elongation complex - IV state

EMDB-22045: 
Mfd-bound E.coli RNA polymerase elongation complex - V state

EMDB-22890: 
Plasmodium falciparum RhopH complex in soluble form

EMDB-21063: 
Density map surrounding actin-like protein in reconstruction of the native gamma-tubulin ring complex

EMDB-22160: 
Structure of SARS-CoV-2 replication-transcription complex bound to nsp13 helicase - nsp13(2)-RTC

EMDB-21911: 
Mdn1-DeltaC alone

EMDB-21912: 
Mdn1-DeltaC plus MIDAS

EMDB-20460: 
Escherichia coli RNA polymerase closed complex (TRPc) with TraR and rpsT P2 promoter

EMDB-20461: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TRPi1) with TraR and rpsT P2 promoter

EMDB-20462: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TRPi1.5a) with TraR and mutant rpsT P2 promoter

EMDB-20463: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TRPi1.5b) with TraR and mutant rpsT P2 promoter

EMDB-20464: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TRPi2) with TraR and rpsT P2 promoter

EMDB-20465: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TpreRPo) with TraR and rpsT P2 promoter

EMDB-20466: 
Escherichia coli RNA polymerase promoter unwinding intermediate (TRPo) with TraR and rpsT P2 promoter
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
