-検索条件
-検索結果
検索 (著者・登録者: cate & j)の結果91件中、1から50件目までを表示しています
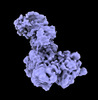
EMDB-40261: 
DDB1/CRBN in complex with ARV-471 and the ER ligand-binding domain
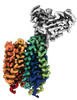
EMDB-43683: 
Cryo-EM structure of FLVCR2 in the inward-facing state with choline bound
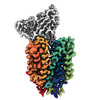
EMDB-43684: 
Cryo-EM structure of FLVCR2 in the outward-facing state with choline bound

PDB-8vzn: 
Cryo-EM structure of FLVCR2 in the inward-facing state with choline bound

PDB-8vzo: 
Cryo-EM structure of FLVCR2 in the outward-facing state with choline bound

EMDB-16963: 
Leishmania tarentolae proteasome 20S subunit in complex with 1-Benzyl-N-(3-(cyclopropylcarbamoyl)phenyl)-6-oxo-1,6-dihydropyridazine-3-carboxamide

PDB-8olu: 
Leishmania tarentolae proteasome 20S subunit in complex with 1-Benzyl-N-(3-(cyclopropylcarbamoyl)phenyl)-6-oxo-1,6-dihydropyridazine-3-carboxamide

EMDB-29449: 
E. coli 70S ribosome with an improved MS2 Tag inserted in H98 (Composite Map)

EMDB-29483: 
E. coli 70S ribosome with an improved MS2 tag inserted in H98 (70S map)

EMDB-29484: 
E. coli 70S ribosome with an improved MS2 tag inserted in H98 (50S focused map)

EMDB-29485: 
E. coli 70S ribosome with an improved MS2 tag inserted in H98 (30S focused map)

PDB-8fto: 
E. coli 70S ribosome with an improved MS2 tag inserted in H98

EMDB-28254: 
Composite 70S ribosome structure for "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates

EMDB-28255: 
70S map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

EMDB-28256: 
30S-focused map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

EMDB-28257: 
50S-focused map for: "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates"

PDB-8emm: 
Composite 70S ribosome structure for "Atomistic simulations of the E. coli ribosome provide selection criteria for translationally active substrates

EMDB-29790: 
30S focus refined map of WT E.coli ribosome complexed with A-site ortho-aminobenzoic acid charged tRNA-Phe

EMDB-29789: 
50S focus refined map of E.coli ribosome complexed with A-site ortho-aminobenzoic acid charged tRNA-Phe

EMDB-29807: 
70S global refined map of WT E.coli ribosome complexed with A-site 3-aminopyridine-4-carboxylic acid charged tRNAPhe

EMDB-29800: 
70S global refined map of E.coli 70S ribosome complexed with A-site meta-aminobenzoic acid charged tRNAPhe

EMDB-29786: 
Structure of WT E.coli 70S ribosome complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site ortho-aminobenzoic acid charged NH-tRNAPhe

EMDB-29788: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site 3-aminopyridine-4-carboxylic acid charged NH-tRNAPhe

PDB-8g6w: 
Structure of WT E.coli 70S ribosome complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site ortho-aminobenzoic acid charged NH-tRNAPhe

PDB-8g6y: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site 3-aminopyridine-4-carboxylic acid charged NH-tRNAPhe

EMDB-29787: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site meta-aminobenzoic acid charged NH-tRNAPhe

PDB-8g6x: 
Structure of WT E.coli ribosome 50S subunit with complexed with mRNA, P-site fMet-NH-tRNAfMet and A-site meta-aminobenzoic acid charged NH-tRNAPhe

EMDB-28165: 
E. coli 70S ribosome with A-loop mutations U2554C and U2555C (Composite Map)

EMDB-28218: 
E. coli 70S ribosome with A-loop mutations U2554C and U2555C (50S Focus Refinement)

EMDB-28229: 
E. coli 70S ribosome with A-loop mutations U2554C and U2555C (30S Focus Refinement)

EMDB-28230: 
E. coli 70S ribosome with A-loop mutations U2554C and U2555C: A-site tRNA class 2

PDB-8eiu: 
E. coli 70S ribosome with A-loop mutations U2554C and U2555C

EMDB-14628: 
SARS-CoV-2 Spike protein in complex with the single chain fragment scFv76

EMDB-14629: 
SARS-CoV-2 Spike RBD in complex with the single chain fragment scFv76 (Focused Refinement)

PDB-7zce: 
SARS-CoV-2 Spike protein in complex with the single chain fragment scFv76

PDB-7zcf: 
SARS-CoV-2 Spike RBD in complex with the single chain fragment scFv76 (Focused Refinement)

EMDB-13967: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 4

PDB-7qh7: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 4

EMDB-13962: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 2

EMDB-13963: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 3

EMDB-13965: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 1

EMDB-13966: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 5

PDB-7qh6: 
Cryo-EM structure of the human mtLSU assembly intermediate upon MRM2 depletion - class 1

EMDB-31536: 
Thermostabilised full length human mGluR5-5M bound with L-quisqualic acid

EMDB-31537: 
Thermostabilised full length human mGluR5-5M with orthosteric antagonist, LY341495

PDB-7fd8: 
Thermostabilised full length human mGluR5-5M bound with L-quisqualic acid

PDB-7fd9: 
Thermostabilised full length human mGluR5-5M with orthosteric antagonist, LY341495

EMDB-23883: 
Single-Particle Cryo-EM Structure of Major Facilitator Superfamily Domain containing 2A in complex with LPC-18:3

PDB-7mjs: 
Single-Particle Cryo-EM Structure of Major Facilitator Superfamily Domain containing 2A in complex with LPC-18:3

EMDB-21966: 
Cryo-EM structure of the GltPh L152C-G321C mutant in the intermediate state.
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
