-検索条件
-検索結果
検索 (著者・登録者: bott & m)の結果196件中、1から50件目までを表示しています

EMDB-16466: 
The structural architecture of alpha-synuclein oligomer

EMDB-16528: 
3D reconstruction of alpha-synuclein oligomer-PSMa3 complex

EMDB-41271: 
Consensus map of 96nm repeat of human respiratory doublet microtubule, RS1-2 region

EMDB-18134: 
Cryo-EM structure of the DNA polymerase holoenzyme E9-A20-D4 of vaccinia virus

EMDB-40811: 
Cryo-EM structure of NLRP3 open octamer
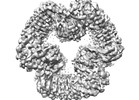
EMDB-40820: 
Cryo-EM structure of NLRP3 closed hexamer
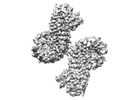
EMDB-40855: 
Structure of NLRP3 and NEK7 complex

PDB-8swf: 
Cryo-EM structure of NLRP3 open octamer

PDB-8swk: 
Cryo-EM structure of NLRP3 closed hexamer

PDB-8sxn: 
Structure of NLRP3 and NEK7 complex

EMDB-16566: 
70S-PHIKZ014 PHIKZ phage protein occupied ribosome

EMDB-40219: 
A subtomogram averaged structure of Helicobacter pylori flagellar motor from a flgV deletion mutant.

EMDB-42149: 
S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004

PDB-8udg: 
S1V2-72 Fab bound to EHA2 from influenza B/Malaysia/2506/2004

EMDB-36891: 
96nm repeat of human respiratory doublet microtubule, IDAf local refined

EMDB-36895: 
Consensus map of 96nm repeat of human respiratory doublet microtubule, RS3 region

EMDB-41299: 
Structural basis of peptidoglycan synthesis by E. coli RodA-PBP2 complex

EMDB-41303: 
Transmembrane map

EMDB-41304: 
Periplasmic map

PDB-8tj3: 
Structural basis of peptidoglycan synthesis by E. coli RodA-PBP2 complex

EMDB-17005: 
Pol I bound to extended and displaced DNA section - closed conformation

EMDB-17033: 
Pol I bound to extended and displaced DNA section - open conformation

PDB-8oo6: 
Pol I bound to extended and displaced DNA section - closed conformation

PDB-8ooy: 
Pol I bound to extended and displaced DNA section - open conformation

EMDB-35888: 
96nm repeat of human respiratory doublet microtubule and associated axonemal complexes

PDB-8j07: 
96nm repeat of human respiratory doublet microtubule and associated axonemal complexes

EMDB-28531: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 in the 3-RBD Down conformation

EMDB-28532: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 with two RBDs in the open conformation

EMDB-28533: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 with two RBDs exposed

PDB-8epn: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 in the 3-RBD Down conformation

PDB-8epp: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 with two RBDs in the open conformation

PDB-8epq: 
Cryo-EM structure of SARS-CoV-2 Spike trimer S2D14 with two RBDs exposed

EMDB-40220: 
96-nm repeat unit of doublet microtubules from Chlamydomonas reinhardtii flagella

PDB-8glv: 
96-nm repeat unit of doublet microtubules from Chlamydomonas reinhardtii flagella

EMDB-15757: 
Structure of RIP2K dimer bound to the XIAP BIR2 domain

PDB-8aza: 
Structure of RIP2K dimer bound to the XIAP BIR2 domain

EMDB-14981: 
Structure of b-galactosidase without reducing agent prepared by Vitrobot

EMDB-14982: 
Structure of b-galactosidase with DTT prepared by Vitrobot

EMDB-14983: 
Structure of b-galactosidase with TCEP prepared by Vitrobot

EMDB-14984: 
Structure of b-galactosidase on continuous carbon support film prepared by Vitrobot

EMDB-15162: 
Structure of the human mitochondrial HSPD1 single ring from grids produced in 14ms

EMDB-15167: 
Structure of the human mitochondrial HSPD1 single ring in the presence of Methionine

EMDB-15176: 
Structure of the human mitochondrial HSPD1 single ring in the presence of ascorbate

EMDB-15178: 
Structure of the human mitochondrial HSPD1 single ring (blotted but no DTT)

EMDB-13604: 
Apo human Kv3.1 cryo-EM structure

EMDB-13605: 
Ligand-bound human Kv3.1 cryo-EM structure (Lu AG00563)

PDB-7pqt: 
Apo human Kv3.1 cryo-EM structure

PDB-7pqu: 
Ligand-bound human Kv3.1 cryo-EM structure (Lu AG00563)

EMDB-12990: 
Cryo-EM structure of N. gonorhoeae LptDE in complex with ProMacrobodies

PDB-7omm: 
Cryo-EM structure of N. gonorhoeae LptDE in complex with ProMacrobodies (MBPs have not been built de novo)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
