-検索条件
-検索結果
検索 (著者・登録者: andrey & z)の結果152件中、1から50件目までを表示しています

EMDB-18687: 
Cryo-EM Structure of Regulated CBS Domain-Containing Pyrophosphatase without One Catalytic Domain

PDB-8skw: 
MicroED structure of d(CGCGCG)2 Z-DNA
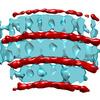
EMDB-16438: 
Toroidal Dps-DNA assembly

EMDB-16439: 
Dps-DNA filament-like assembly
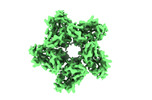
EMDB-35143: 
Cryo-EM structure of the zeaxanthin-bound kin4B8

PDB-8i2z: 
Cryo-EM structure of the zeaxanthin-bound kin4B8

EMDB-28197: 
Escherichia coli 70S ribosome bound to thermorubin, deacylated P-site tRNAfMet and aminoacylated A-site Phe-tRNA

PDB-8ekc: 
Escherichia coli 70S ribosome bound to thermorubin, deacylated P-site tRNAfMet and aminoacylated A-site Phe-tRNA

EMDB-13485: 
Cryo-EM structure of Bestrhodopsin (rhodopsin-rhodopsin-bestrophin) complex

PDB-7pl9: 
Cryo-EM structure of Bestrhodopsin (rhodopsin-rhodopsin-bestrophin) complex

EMDB-14943: 
Structure of DPS

EMDB-14948: 
Dps nanocage with the "small circle" mineral core

EMDB-14949: 
Dps nanocage with the "dumbbell" mineral core

EMDB-14950: 
Dps nanocage with a medium mineral core

EMDB-14951: 
Dps nanocage with a large mineral core

EMDB-14952: 
Dps nanocage with the symmetrical hollow mineral core

EMDB-13741: 
Structure of Candida albicans 80S ribosome in complex with cycloheximide

EMDB-13750: 
Cryo-EM map of the Candida albicans 80S ribosome in complex with blasticidin s

PDB-7q08: 
Structure of Candida albicans 80S ribosome in complex with cycloheximide

PDB-7q0r: 
Structure of the Candida albicans 80S ribosome in complex with blasticidin s

EMDB-13737: 
Cryo-EM map of the vacant Candida albicans 80S ribosome

EMDB-13744: 
Cryo-EM map of Candida albicans 80S ribosome in complex with phyllanthoside

EMDB-13749: 
Cryo-EM map of the Candida albicans 80S ribosome in complex with anisomycin

PDB-7pzy: 
Structure of the vacant Candida albicans 80S ribosome

PDB-7q0f: 
Structure of Candida albicans 80S ribosome in complex with phyllanthoside

PDB-7q0p: 
Structure of the Candida albicans 80S ribosome in complex with anisomycin

EMDB-13389: 
human 20S proteasome (before post-processing)

PDB-7pg9: 
human 20S proteasome

EMDB-31724: 
20S+monoUb-CyclinB1-NT (S1)

EMDB-31727: 
20S+monoUb-CyclinB1-NT (S2)

EMDB-31728: 
20S proteasome incubated with monoUb-CyclinB1-NT (S0)

EMDB-31730: 
20S proteasome (after post-processing)

PDB-7v5g: 
20S+monoUb-CyclinB1-NT (S1)

PDB-7v5m: 
20S+monoUb-CyclinB1-NT (S2)

EMDB-24404: 
Covalent inhibition of hAChE by organophosphates causes homodimer dissociation through long-range allosteric effects

EMDB-13017: 
RqcH DR variant bound to 50S-peptidyl-tRNA-RqcP RQC complex (rigid body refinement)

PDB-7ope: 
RqcH DR variant bound to 50S-peptidyl-tRNA-RqcP RQC complex (rigid body refinement)

EMDB-12961: 
Dps with iron-containing cluster

EMDB-11978: 
Nanobody E bound to Spike-RBD in a localized reconstruction

EMDB-11981: 
SARS-CoV-spike bound to two neutralising nanobodies

PDB-7b14: 
Nanobody E bound to Spike-RBD in a localized reconstruction

PDB-7b18: 
SARS-CoV-spike bound to two neutralising nanobodies

EMDB-11980: 
SARS-CoV-spike RBD bound to two neutralising nanobodies.

PDB-7b17: 
SARS-CoV-spike RBD bound to two neutralising nanobodies.
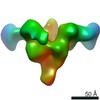
EMDB-22054: 
Negative stain EM map of SUDV GPdMuc in complex with EA97

EMDB-22055: 
Negative stain EM map of SUDV GPdMuc in complex with EB46 Fab

EMDB-23018: 
SARS-CoV-2 spike in complex with nanobodies E

PDB-7ksg: 
SARS-CoV-2 spike in complex with nanobodies E

PDB-7khe: 
Escherichia coli RNA polymerase and rrnBP1 promoter pre-open complex with DksA/ppGpp

EMDB-11616: 
Cryo-EM structure of the SARS-CoV-2 spike protein bound to neutralizing sybodies (Sb23)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
