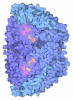
登録情報 データベース : PDB / ID : 5n2aタイトル METHYL-COENZYME M REDUCTASE III FROM METHANOTORRIS FORMICICUS TRIGONAL FORM (Methyl-coenzyme M reductase, ... Methyl-coenzyme M reductase subunit alpha キーワード / / / / / / / / / / / / / / / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Methanotorris formicicus Mc-S-70 (古細菌)手法 / / / 解像度 : 2.8 Å データ登録者 Wagner, T. / Wegner, C.E. / Ermler, U. / Shima, S. ジャーナル : J.Bacteriol. / 年 : 2017タイトル : Phylogenetic and Structural Comparisons of the Three Types of Methyl Coenzyme M Reductase from Methanococcales and Methanobacteriales.著者 : Wagner, T. / Wegner, C.E. / Kahnt, J. / Ermler, U. / Shima, S. 履歴 登録 2017年2月7日 登録サイト / 処理サイト 改定 1.0 2017年6月14日 Provider / タイプ 改定 2.0 2019年4月24日 Group / Data collection / Database references / カテゴリ / citation / pdbx_database_procItem _atom_site.occupancy / _citation.journal_abbrev ... _atom_site.occupancy / _citation.journal_abbrev / _citation.journal_volume / _citation.title 改定 2.1 2024年1月17日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn / struct_conn_type Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr2_auth_comp_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_atom_id / _pdbx_struct_conn_angle.ptnr2_label_comp_id / _pdbx_struct_conn_angle.ptnr2_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_conn_type.id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード TRANSFERASE (転移酵素) /
TRANSFERASE (転移酵素) /  POST-TRANSLATIONAL MODIFICATION (翻訳後修飾) /
POST-TRANSLATIONAL MODIFICATION (翻訳後修飾) /  BINDING SITES (結合部位) /
BINDING SITES (結合部位) /  CATALYSIS /
CATALYSIS /  COENZYMES (補因子) /
COENZYMES (補因子) /  DISULFIDES (ジスルフィド) /
DISULFIDES (ジスルフィド) /  HYDROGEN (水素) /
HYDROGEN (水素) /  HYDROGEN BONDING (水素結合) /
HYDROGEN BONDING (水素結合) /  LIGANDS (リガンド) /
LIGANDS (リガンド) /  MESNA (メスナ) /
MESNA (メスナ) /  METALLOPORPHYRINS /
METALLOPORPHYRINS /  METHANE (メタン) /
METHANE (メタン) /  METHANOCOCCALES /
METHANOCOCCALES /  NICKEL (ニッケル) /
NICKEL (ニッケル) /  OXIDATION-REDUCTION (酸化還元反応) /
OXIDATION-REDUCTION (酸化還元反応) /  OXIDOREDUCTASES (酸化還元酵素) /
OXIDOREDUCTASES (酸化還元酵素) /  PHOSPHOTHREONINE (トレオニン) / PROTEIN CONFORMATION /
PHOSPHOTHREONINE (トレオニン) / PROTEIN CONFORMATION /  PROTEIN FOLDING (フォールディング) /
PROTEIN FOLDING (フォールディング) /  PROTEIN STRUCTURE (タンパク質構造) /
PROTEIN STRUCTURE (タンパク質構造) /  THERMOPHILE (好熱菌) /
THERMOPHILE (好熱菌) /  AUTOTROPH (独立栄養生物) / HYDROXY-TRYPTOPHANE
AUTOTROPH (独立栄養生物) / HYDROXY-TRYPTOPHANE 機能・相同性情報
機能・相同性情報 coenzyme-B sulfoethylthiotransferase /
coenzyme-B sulfoethylthiotransferase /  coenzyme-B sulfoethylthiotransferase activity /
coenzyme-B sulfoethylthiotransferase activity /  メタン生成経路 /
メタン生成経路 /  metal ion binding
metal ion binding
 Methanotorris formicicus Mc-S-70 (古細菌)
Methanotorris formicicus Mc-S-70 (古細菌) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.8 Å
分子置換 / 解像度: 2.8 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Bacteriol. / 年: 2017
ジャーナル: J.Bacteriol. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5n2a.cif.gz
5n2a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5n2a.ent.gz
pdb5n2a.ent.gz PDB形式
PDB形式 5n2a.json.gz
5n2a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/n2/5n2a
https://data.pdbj.org/pub/pdb/validation_reports/n2/5n2a ftp://data.pdbj.org/pub/pdb/validation_reports/n2/5n2a
ftp://data.pdbj.org/pub/pdb/validation_reports/n2/5n2a リンク
リンク 集合体
集合体

 要素
要素 Coenzyme-B sulfoethylthiotransferase
Coenzyme-B sulfoethylthiotransferase
 Methanotorris formicicus Mc-S-70 (古細菌)
Methanotorris formicicus Mc-S-70 (古細菌) coenzyme-B sulfoethylthiotransferase
coenzyme-B sulfoethylthiotransferase Coenzyme-B sulfoethylthiotransferase
Coenzyme-B sulfoethylthiotransferase
 Methanotorris formicicus Mc-S-70 (古細菌)
Methanotorris formicicus Mc-S-70 (古細菌) coenzyme-B sulfoethylthiotransferase
coenzyme-B sulfoethylthiotransferase Coenzyme-B sulfoethylthiotransferase
Coenzyme-B sulfoethylthiotransferase
 Methanotorris formicicus Mc-S-70 (古細菌)
Methanotorris formicicus Mc-S-70 (古細菌) coenzyme-B sulfoethylthiotransferase
coenzyme-B sulfoethylthiotransferase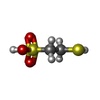

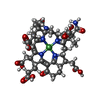






 補酵素B
補酵素B F430
F430 臭化物
臭化物 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X10SA / 波長: 0.99998 Å
/ ビームライン: X10SA / 波長: 0.99998 Å : 0.99998 Å / 相対比: 1
: 0.99998 Å / 相対比: 1  解析
解析 :
:  分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj